中部大学
自然科学研究機構 基礎生物学研究所
筑波大学
1. 研究成果のポイント
-
約2100万個のマウスの単一細胞遺伝子発現データセット「mouse-Genecorpus-20M」を構築し、Transformer Encoderアーキテクチャを用いて事前学習したマウス版の大規模基盤モデル(注1)「Mouse-Geneformer」を開発。
-
Mouse-Geneformerをファインチューニングすることで、コンピュータ上での遺伝子操作シミュレーション実験(in silico遺伝子摂動実験(注2))を実現し、動物による実験と同等の結果が得られた。これにより、動物実験のコストを大幅に削減できる可能性を示した。
-
相同遺伝子(注3)変換することで、Mouse-Geneformerを用いてヒトのデータの解析も可能である事を示した。
-
大規模基盤モデルを用いた異種間トランスクリプトーム解析(注4)の有用性を示し、創薬研究や進化生物学への貢献、さらには非モデル生物への応用の可能性も提示。
2. 発表概要
近年、生成系AI技術の発展により、ChatGPTなどに代表される大規模基盤モデルが様々な技術分野に急速に利用が普及しつつあります。遺伝子解析の研究分野においても、2023年に米国の研究グループが発表したヒト単一細胞遺伝子発現データを大量に学習した「Geneformer」(注1、注6)が注目を集めました。この技術により、細胞の遺伝子発現データから高精度に細胞型を分類することや、細胞内の遺伝子発現変動をコンピューター上でシミュレーションすることが可能になりました。
一方で、基礎研究の現場では、マウスが重要なモデル生物として広く利用されており、膨大な量の遺伝子発現データが蓄積されています。このデータを活用し人工知能(AI)で細胞の変異や異常を予測し、さらにヒトに応用することができれば、創薬や医療が大きく前進することが期待されます。
このたび、中部大学工学部情報工学科の山下隆義教授、藤吉弘亘教授、伊藤啓太大学院生、基礎生物学研究所 超階層生物学センターおよび筑波大学生存ダイナミクス研究センター(TARA)の重信秀治教授(クロスアポイントメント)らの研究グループは、マウス版Geneformerである「Mouse-Geneformer」の開発に成功しました。本モデルは、先行研究であるヒト版Geneformerを基盤に、最先端のAI技術であるTransformer Encoderアーキテクチャを用いて2100万細胞のマウス単一細胞遺伝子発現データを事前学習させた、遺伝子版大規模基盤モデルです。
研究グループは、Mouse-Geneformerを用いて、遺伝子発現データからのマウス細胞の細胞型分類や、細胞内の遺伝子発現変化の予測が高精度に可能であることを実証しました。従来手法と比較し、細胞型分類の精度が向上しました。また、in silico遺伝子摂動実験によって疾患に関連すると考えられる遺伝子を特定できることが示されました。さらに、研究グループは本モデルを異種間解析に応用する手法も提案しました。相同遺伝子
(注3)変換を介してヒトのデータをMouse-Geneformerで解析すると、ヒト版Geneformerと同等の精度でヒトの細胞型分類が可能であることを確認しました。マウスの遺伝子発現データは、ヒトよりも多く収集・公開されており、また、ヒトでは技術的・倫理的に困難な実験のデータも取得可能です。このため、Mouse-Geneformerと異種間解析技術を組み合わせることによって、創薬や疾患研究の基盤情報として大きな貢献が期待されます。本研究成果は、3月19日付で、遺伝学の国際誌PLOS Geneticsに掲載されました。
3. 研究の背景
単一細胞RNAシーケンス(注5)は、個々の細胞レベルで遺伝子発現プロファイルを定量化する強力な技術であり、これから得られる単一細胞遺伝子発現データは、細胞の多様性や発生過程の詳細な理解を大きく促進しています。しかし、このような大規模な単一細胞遺伝子発現データから細胞の多様性や疾患メカニズムに関する知見を抽出するためには、高度な計算手法が求められます。そのため、深層学習の技術を用いた単一細胞遺伝子発現データの解析手法の開発が進んでいます。特に、Transformer Encoderアーキテクチャを基盤とするGeneformer(注1、注6)は、ヒトの単一細胞遺伝子発現データを大量に用いた事前学習により、ヒト細胞において細胞の遺伝子発現データから高精度に細胞型を分類することや、細胞内の遺伝子発現変動をコンピューター上でシミュレーションすることが可能になりました。
マウス(
Mus musculus)は、生物学および医学研究において最も広く利用されている重要なモデル生物であり、その遺伝的背景や生理学的特性に関する膨大実験データとともに、数多くの単一細胞遺伝子発現データがデータベースに蓄積されています。しかしながら、既存のGeneformerはヒトの単一細胞遺伝子発現データを用いて事前学習しているため、マウスの単一細胞遺伝子発現データの解析には適用が難しいという課題がありました。そこで本研究では、大規模なマウスの単一細胞遺伝子発現データセット「mouse-Genecorpus-20M」を構築し、Geneformerアーキテクチャを事前学習することで、マウスの単一細胞遺伝子発現データを解析可能な深層学習モデル「Mouse-Geneformer」を開発しました。
4. 研究の成果
本研究では、マウス固有の遺伝子ネットワークを反映したGeneformerモデルの開発を目的とし、公的データベース(PanglaoDB、Single Cell Expression Atlas、Single Cell Portal、ENCODE project、10x Genomics、CELLxGENE)から年齢や臓器が多様に異なる単一細胞遺伝子発現データを収集・統合しました。その結果、約2100万個の単一細胞データから構成される大規模データセット「mouse-Genecorpus-20M」を構築しました(図1)。このデータセットを用いてTransformer EncoderアーキテクチャのGeneformerモデルにマウスの遺伝子ネットワークを事前学習させることで「Mouse-Geneformer」を開発しました(図2)。開発したMouse-Geneformerは従来手法(scDeepSort, scVAE)と比較して、遺伝子発現データからのマウス細胞の細胞型分類において高い精度を示しました。特に、乳腺や四肢の筋肉、心臓、脳などの分類精度が大幅に向上し、モデルの有効性が確認されました(図3)。また、腎臓病の疾患マウスモデルとCop1遺伝子をノックアウトした疾患マウスモデルを用いてin silico遺伝子摂動実験(遺伝子の発現量を変化させてコンピュータに入力するシミュレート実験)を実施したところ、疾患関連遺伝子の特定が可能であることや、Mouse-Geneformerが疾患状態を分類できることも示されました(図4)。
さらに本研究では、Mouse-Geneformerの異種間解析への応用も検討しました。ヒトの遺伝子をマウスの遺伝子に相同遺伝子変換し、Mouse-Geneformerでヒトの単一細胞遺伝子発現データの細胞型分類およびin silico遺伝子摂動実験を行った結果、細胞型分類ではMouse-Geneformerはヒト版Geneformerと同等の分類精度が得られました。また、ヒトの心筋梗塞に関連する遺伝子をマウスの遺伝子に相同遺伝子変換してin silico遺伝子摂動実験を行った場合でも、ヒト版Geneformerと同様の疾患関連遺伝子が特定できました。一方で、同様の相同遺伝子変換による実験をCOVID-19で行ったところ、ヒト版Geneformerで特定できたCOVID-19に関連する遺伝子群を、マウス版Geneformerでは十分には特定できず、マウスとヒトの間における種特異的な遺伝子ネットワークの違いが影響していることが示唆されました。これは、マウスがSARS-CoV-2(新型コロナウイルス)に自然感染しにくいという生物学的背景と一致しており、同時に種特異的なGeneformerモデルを構築することの意義と重要性も示していると言えます。
なお、本研究で構築したMouse-Genecourpus-20M データセットとMouse-Geneformer モデルは、オープンソースブラットフォームであるHugging FaceとGitHubにて公開しています。(
MPRG/Mouse-Genecorpus-20M · Datasets at Hugging Face:
GitHub - machine-perception-robotics-group/Mouse-Geneformer)
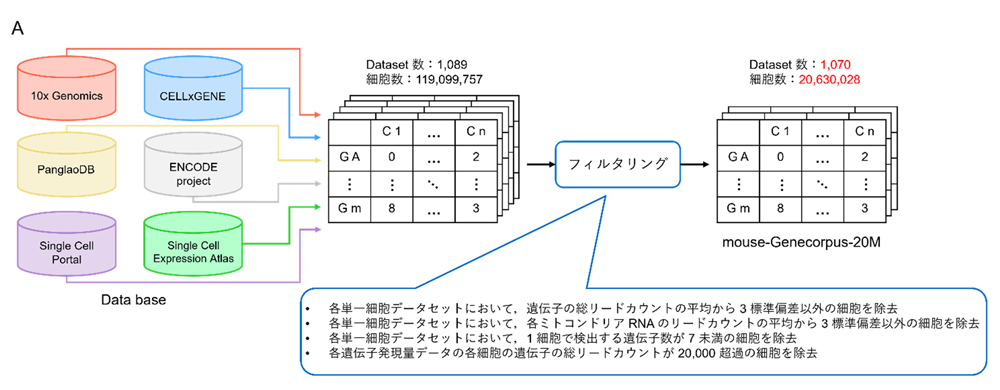
図1:マウスの大規模単一細胞遺伝子発現データセット「Mouse-Genecorpus-20M」を構築する手順
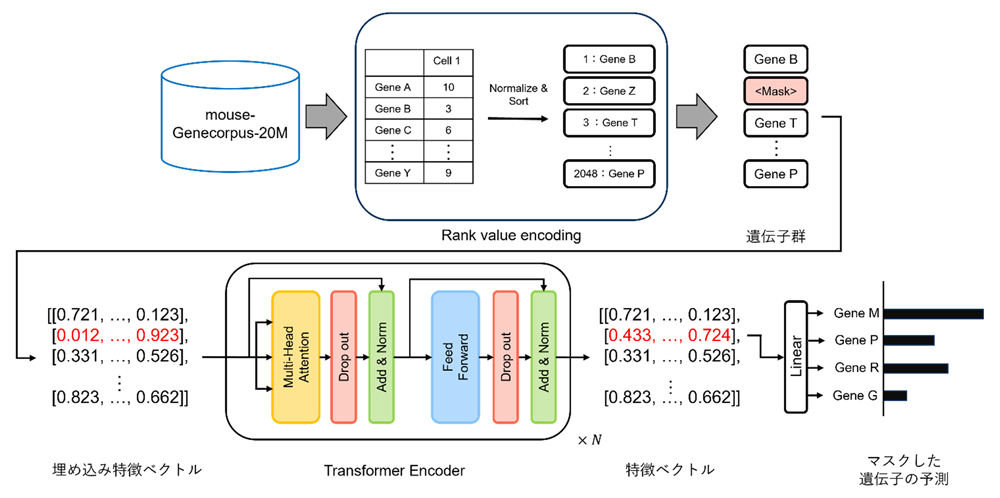
図2:Mouse-Genecorpus-20MをTransformerアーキテクチャによって深層学習して「Mouse-Geneformer」を構築する概略図。
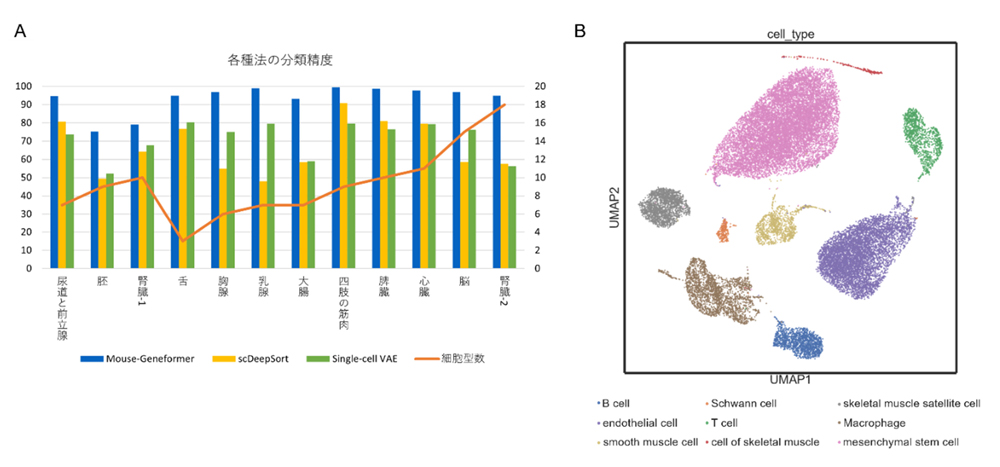
図3:A)各種法の細胞型分類精度とB)四肢の筋肉の特徴ベクトルの可視化結果
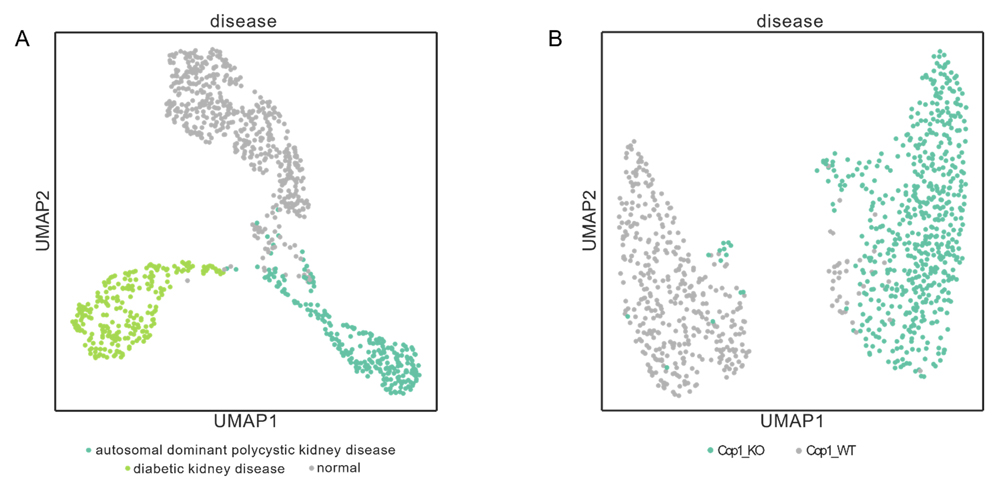
図4:A)腎臓病のマウスモデルの特徴ベクトルの可視化結果とB)Cop1遺伝子をノックアウトした疾患マウスモデルの特徴ベクトルの可視化結果
5. 今後の展望
本研究により、Mouse-Geneformerがマウスの単一細胞遺伝子発現データ解析において高精度な細胞型分類および疾患関連遺伝子の同定に有効であることが示されました。本研究で構築した「mouse-Genecorpus-20M」や「Mouse-Geneformer」は、深層学習を活用した解析モデルの基盤として機能し、マウスの遺伝学や疾患モデルの理解を加速度的に進展させると期待されます。特に、マウスモデルを用いた疾患研究では、in silico遺伝子摂動実験により、疾患原因の候補遺伝子や治療標的の探索がコンピュータ上で可能となり、創薬や治療戦略の確立に貢献できると考えられます。
また、Mouse-Geneformerのヒト研究への応用の可能性も示され、特に倫理的・技術的制約によってサンプル採取が難しいヒト胎児組織や特定の疾患モデルの研究に貢献できると期待されます。さらに、本研究で提案した異種間解析手法は、ヒトやマウスに限らず、他の生物種にも応用可能と考えられ、特に大規模な遺伝子発現データを取得しにくい生物種を対象とする研究への展開も期待されます。今後は、データのさらなる拡充や解析手法の高度化を通じて、創薬や医療、進化生物学など幅広い領域への応用が見込まれます。
6. 論文の情報
雑誌名: PLOS Genetics
論文タイトル:Mouse-Geneformer: A Deep Learning Model for Mouse Single-Cell Transcriptome and Its Cross-Species Utility
著者:Keita Ito, Tsubasa Hirakawa, Shuji Shigenobu*, Hironobu Fujiyoshi*, Takayoshi Yamashita*(*責任著者)
DOI: 10.1371/journal.pgen.1011420
URL:
https://doi.org/10.1371/journal.pgen.1011420
研究費:基礎生物学研究所統合ゲノミクス共同利用研究(24NIBB462)
備考:本研究は中部大学と基礎生物学研究所との研究、教育等の連携に関する包括協定を通して行われた。また、自然科学研究機構2023年度OPEN MIX LAB公募研究プログラム(課題番号OML042301)の支援により開催された合同ワークショップが本共同研究の契機となった。
7. 用語説明
-
注1 大規模基盤モデル:英語ではFoundation Modelと言う。Fundation Model の1つがインターネットサービスの生成AIや対話型生成AIに用いる大規模言語モデル(Large Language Model:LLM)である。対話型AIの場合、ネット上の大量のテキストデータを学習したLLMが次に来る単語を用いて文章を作る。言語でなく遺伝子情報をテキストとみなして学習すれば細胞の振る舞いを予測できる。2023年に米マサチューセッツ工科大学(MIT)とハーバード大学の共同研究チームがヒトの遺伝子情報を用いる基盤モデルの「Geneformer」を発表した。
-
注2 in silico:生物学や医学の研究で用いられる用語の一つで、コンピュータを用いたシミュレーションやデータ解析を指す言葉。日本語ではイン・シリコと記載する。このほか試験管内での実験をin vitro(イン・ビトロ)、生体内の本来の場所での実験や解析をsitu(イン・シチュー)と言う。
-
注3 相同遺伝子(ホモログ):共通祖先から進化した遺伝子を相同遺伝子(ホモログ)と呼ぶ。ゲノム進化の議論や遺伝子機能の推定において重要な手がかりとなる。ホモログはさらにオーソログとパラログに分類される。今回の解析ではオーソログの関係に注目して遺伝子変換を行った。オーソログとは共通の祖先遺伝子から種分岐に伴って派生した遺伝子間の対応関係、または対応関係にある遺伝子群を指す。異なる生物種のオーソログ間で遺伝子の機能は保存されていると考えられている。
-
注4 トランスクリプトーム解析:細胞や組織などにおいて転写されたRNA(リボ核酸)全体をトランスクリプトーム、それを網羅的に解析するのをトランスクリプトーム解析と言う。ゲノムが全ての細胞でほぼ同一なのに対し、トランスクリプトームは細胞の機能に応じた転写や転写後の調節を反映しており、細胞や遺伝子の機能についての情報を得ることができる。
-
注5 単一細胞RNAシーケンス:シングルセルRNA-seq解析(scRNA-seq)とも呼ばれる。RNAシーケンス(RNA-seq)は生物の個体や組織などの網羅的遺伝子発現(トランスクリプトーム)を次世代シーケンサーで網羅的に解析する技術であるが、これを単一細胞ごとに行うことで、個々の細胞レベルでの遺伝子発現プロファイルを取得できる。近年、単一細胞レベルでのRNAシーケンスを超並列かつ効率的に実施する技術が開発され、大量のシングルセルRNA-seqデータが産出されるようになった。
-
注6 ヒト版Geneformer: .Theodoris CV, Xiao L, Chopra A, Chaffin MD, Al Sayed ZR, Hill MC, et al. Transfer learning enables predictions in network biology. Nature. 2023;618(7965):616–24. doi: 10.1038/s41586-023-06139-9.
8. お問い合わせ先
【研究内容について】
山下隆義 中部大学 工学部情報工学科 教授
重信 秀治 基礎生物学研究所 超階層生物学センター・進化ゲノミクス研究室 教授・筑波大学 生存ダイナミクス研究センター(TARA)教授
電子メール shige@nibb.ac.jp
電話 0564-55-7670
【報道担当】
中部大学
入試・広報センター
電子メール chubu-info@fsc.chubu.ac.jp
電話 0568-51-7638
基礎生物学研究所 広報室
電子メール press@nibb.ac.jp
電話 0564-55-7628
FAX 0564-55-7597
筑波大学 広報局
電子メール kohositu@un.tsukuba.ac.jp
電話 029-853-2040