ヒトの体は、200種類以上の細胞から構成され、各細胞での固有の性質を持つように、細胞が遺伝子の使い方を変え、必要に応じて遺伝子のスイッチをON、OFFすることにより調節しています。実際に種々の細胞遺伝子の働き方を調べてみると、約10%程度の遺伝子しかONになっておらず、残りの90%の遺伝子はOFFになっています。また、がん細胞ではこのOFFの状態をきちんと維持できないことが知られています。遺伝子のスイッチがOFFになった状態の代表的なマークのひとつとして、ヒストンH3と呼ばれるタンパク質の9番目のリシンのメチル化修飾(H3K9メチル化)が挙げられますが、このH3K9メチル化がどのように制御されているのかよくわかっていませんでした。
今回、基礎生物学研究所クロマチン制御研究部門の中山潤一教授、名古屋市立大学の大屋恵梨子研究員(現 中央大学助教)らは、分裂酵母を用いて、H3K9のメチル化修飾がヒストンH3の14番目のリシンのユビキチン化修飾によって促進されることを明らかにしました。この成果により、遺伝子をOFFにする仕組みのメカニズムの一端が明らかになりました。本研究は、2019年8月29日に欧州の科学誌「EMBO reports」に掲載されました。
【研究の背景】
ヒトの体は、ひとつの受精卵が細胞分裂を繰り返し、200種類以上の細胞に分かれていくことで形成されます。細胞分裂では正確にDNAがコピーされるため、これらの細胞は基本的に同じ遺伝子のセットを持っていますが、脳の神経細胞や筋肉の細胞、皮膚の細胞というように、個々の細胞の形や機能は異なります。これは、細胞が分化する過程において、組み合わせの異なる遺伝子のスイッチをON、あるいはOFFにすることで、固有の性質を持つように遺伝子の使い方を変えているためです。
この遺伝子のスイッチングには、DNAの塩基配列として書き込まれた遺伝情報の変化は伴わず、DNAとそれを巻き取る糸巻きのような働き方をするヒストンと呼ばれるタンパク質との複合体(クロマチン)への化学修飾によって調節されています。たとえば、ヒストンH3の4番目のリシンのメチル化や、9番目のリシンがアセチル化されると、クロマチンが開いた状態(ユークロマチン)になり、遺伝子の発現が促進されますが、ヒストンH3の9番目のリシンがメチル化されると、クロマチンが高度に凝縮し、遺伝子発現が抑制されます(ヘテロクロマチン)。これまでの研究によって、ヒストンの修飾と遺伝子発現の関係は明らかにされてきましたが、それぞれの修飾がどのように制御されているのかについての詳細はよくわかっていませんでした。
本研究では、遺伝子発現の発現調節のメカニズムが私たちヒトとよく似ている分裂酵母をモデル生物として用いました。分裂酵母のヒストンH3の9番目のリシン(H3K9)をメチル化する酵素Clr4(ヒトSUV39H1/2のホモログ)は、細胞内で他のタンパク質と複合体を形成しており、この複合体はClr4メチル化酵素複合体(CLRC)と呼ばれています。その複合体の中には、ユビキチン化修飾を結合させる酵素として知られているCullin(Cul4)が含まれており、このCul4によるユビキチン化修飾が抑制的なヘテロクロマチンの形成に関わっていることが示唆されていました。しかし、このCul4がどのタンパク質をユビキチン化するのか、その修飾がどのようにヘテロクロマチン形成に関わるのか長い間不明のままでした。
【研究の成果】
本研究では、まずCLRC複合体の精製を行い、質量分析法によりCLRCと複合体を形成するタンパク質を探索した結果、CLRCがヒストンと相互作用することを見出しました。次に、ヒストンがCLRCに含まれるCul4によってユビキチン化されるかどうか試験管内で調査したところ、4種類のヒストンのうち、ヒストンH3が特異的にユビキチン化修飾を受けることが分かりました。次に、ヒストンH3のどのアミノ酸残基がユビキチン化修飾を受けるかどうか質量分析法により確認したところ、14番目のリシンが優先的にユビキチン化修飾を受けることを見出しました。
ここまで試験管内の人工的な系で確認してきましたが、これまで分裂酵母においてヒストンH3の14番目のリシン(H3K14)がユビキチン化修飾を受けるという報告はなく、実際に生体内で同様のことが起こっているか確認することにしました。CLRCがヘテロクロマチン領域に局在しているという過去の知見を踏まえ、ヘテロクロマチン領域のクロマチン免疫沈降法し質量分析法により検証したところ、H3K14のユビキチン化修飾はヘテロクロマチン領域に多く局在しているということが分かりました。
さらに、H3K14とヘテロクロマチン構造との関係を調査しました。ヒストンH3の14番目のリシンを他のアミノ酸に置換したところ、ヘテロクロマチンのマークであるH3K9のメチル化修飾が減少し、遺伝子の発現抑制が出来なくなったことから、H3K14はヘテロクロマチン形成に重要であることが示唆されました。
それでは、ヒストンH3K14のユビキチン化修飾が、ヘテロクロマチン形成にどのように影響するのでしょうか。私たちは、H3K14のユビキチン化がH3K9のメチル化に何らかの影響を与えるのではないかという仮説を立て、未修飾のヒストンH3と14番目のリシンがユビキチン化されたヒストンH3をそれぞれ基質として用い、Clr4によるメチル化アッセイを行いました。興味深いことに、Clr4は未修飾のヒストンH3に比べて、14番目のリシンがユビキチン化されたヒストンH3を約6倍も効率よくメチル化することが分かりました。
この結果は、Clr4がユビキチン化されたヒストンH3を認識して結合するという事を示唆しています。Clr4のどの領域でユビキチンを認識し、結合しているのか検証した結果、Clr4のアミノ末端側の領域がユビキチン化されたヒストンH3との結合に必要であることが示唆されました。
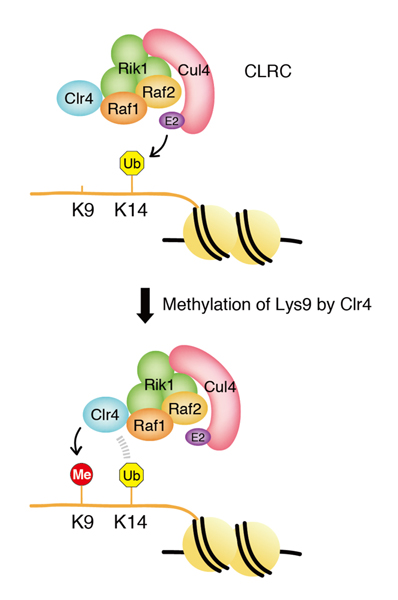
以上の結果より、CLRCによるH3K14のユビキチン化修飾はClr4によって認識され、ヘテロクロマチンの代表的なマークであるH3K9のメチル化修飾を促進し、ヘテロクロマチン形成に寄与していることが明らかになりました(図1)。
図1.CLRCによるヒストンH3K14ユビキチン化はClr4によるH3K9メチル化を促進する。
【今後の展望】
本研究成果によって、遺伝子をOFFにする仕組みに関与する新しい修飾が明らかになりました。この修飾を糸口として、遺伝子発現の調節メカニズムの全貌が明らかになることが期待されます。さらに、近年DNAやヒストンの修飾異常を標的とした治療が臨床試験の段階にあり、新しく見出された修飾と遺伝子の発現制御の関係を明らかにすることは、がんなどの疾患の理解、有効な診断や治療標的としての展望が期待できます。
【発表雑誌】
雑誌名: EMBO reports
掲載日: 2019年8月29日
論文タイトル:H3K14 ubiquitylation promotes H3K9 methylation for heterochromatin assembly.
著者:Eriko Oya
1,2, Reiko Nakagawa
3, Yuriko Yoshimura
4, Mayo Tanaka
4, Gohei Nishibuchi
1 ,Shinichi Machida
5, Atsuko Shirai
6, Karl Ekwall
2, Hitoshi Kurumizaka
5,7,Hideaki Tagami
1, Jun-ichi Nakayama
1,4,8
(
1名古屋市立大学システム自然科学研究科,
2Department of Biosciences and Nutrition, Karolinska Institutet, Sweden,
3理化学研究所CDB系統情報学研究室,
4基礎生物学研究所クロマチン制御研究部門,
5早稲田大学大学院先進理工学研究科,
6理化学研究所細胞記憶研究室,
7東京大学定量生命科学研究所,
8総合研究大学院大学生命科学研究科)
DOI: 10.15252/embr.201948111 (
https://doi.org/10.15252/embr.201948111)
【研究グループ】
基礎生物学研究所クロマチン制御研究部門の中山潤一教授を中心として、名古屋市立大学の田上英明准教授、早稲田大学の胡桃坂仁志教授(現東京大学)、カロリンスカ研究所(スウェーデン)のKarl Ekwall教授との共同研究として実施されました。
【研究サポート】
本研究は以下の支援を受けて行われました。
文部科学省科学研究費助成事業(JP23114005, JP26291072, JP17H03713, JP18H05532, JP18H05534)
持田記念医学薬学振興財団
上原記念生命科学財団
武田科学振興財団
The Swedish Cancer Society
The Swedish Research Council
【本研究に関するお問い合わせ先】
基礎生物学研究所 クロマチン制御研究部門
教授 中山 潤一(なかやま じゅんいち)
TEL: 0564-55-7680
E-mail: jnakayam@nibb.ac.jp
【報道担当】
基礎生物学研究所 広報室
TEL: 0564-55-7628
FAX: 0564-55-7597
E-mail: press@nibb.ac.jp