自然科学研究機構 基礎生物学研究所
科学技術振興機構(JST)
アーバスキュラー菌根菌
注1(以下AM菌)は植物の根の中に菌糸を発達させるとともに、土中にも菌糸を張り巡らし、植物の根が届かない場所のリン等を植物に届け、代わりに糖などの光合成産物を受け取る共生関係を築いています。この共生関係は植物の生育を促進する効果があり、将来的に、リン肥料などの消費を押さえる生物資材としての活用が期待されています(図1)。
今回、基礎生物学研究所の前田太郎研究員、重信秀治特任准教授、川口正代司教授らを中心とした研究グループは、代表的なAM菌である
Rhizophagus irregularis(図2)のゲノムを従来よりも格段に高精度に解読することに成功し、AM菌が脂肪酸やビタミンB1などの栄養素の合成に関わる遺伝子を欠損していることを明確にしました。AM菌は植物との共生無しでは胞子増殖できませんが、今後これらの栄養素を人為的に投与することによってAM菌を単独で大量培養できる可能性があります。さらに、AM菌の種の同定や植物への効果の評価に使われてきた遺伝子マーカー
注2であるリボソームDNA遺伝子
注3が、他の生物では見られない特殊な性質をもつことを明らかにしました。
本研究成果は「Communications Biology」に2018年7月10日付けで掲載されます。
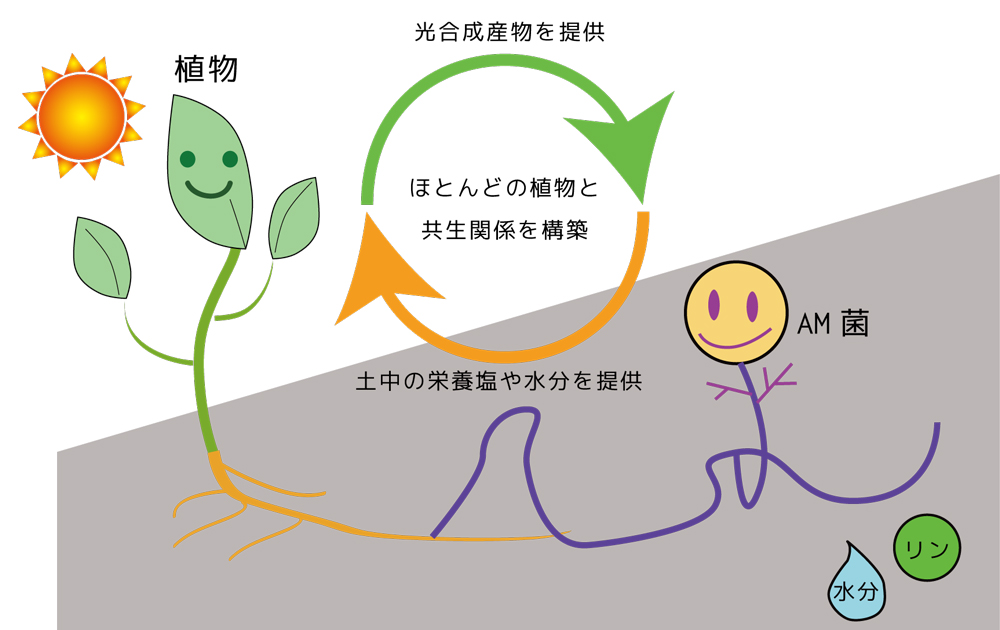 図1 アーバスキュラー菌根菌と植物の共生現象の概念図
図1 アーバスキュラー菌根菌と植物の共生現象の概念図
アーバスキュラー菌根菌は、植物の根の中に菌糸を発達させると同時に土壌中にも菌糸を張り巡らし、リンやミネラル、水分を宿主植物に届ける役割をする。一方、植物は光合成によって生産された糖や脂肪酸をAM菌に与える。この共生はほとんどの植物で見られ、陸上生態系に重要な役割を果たしている。
 図2 アーバスキュラー菌根菌と植物根
図2 アーバスキュラー菌根菌と植物根
植物の根(ニンジン毛状根)とそれに共生しているAM菌
Rhizophagus irregularis DAOM181602株の実体顕微鏡像
【研究の背景】
植物の根には多くの真菌や細菌が共生しており、植物の成長や陸上生態系において重要な役割を果たしています。アーバスキュラー菌根菌は植物と共生する真菌類の中でも最も歴史が古く、植物が海から陸に進出した4億年以上前には、すでに共生していたと考えられています。AM菌は宿主植物の根に菌糸を発達させると同時に、土中にも菌糸を張り巡らし、植物の根が届かない場所のリンやミネラルを効率よく吸収し植物に届けます。代わりに、植物は糖などの光合成産物をAM菌に与えることで共生関係を成立させています(図1)。この古くから続く共生関係は、陸上生態系に重要な役割を果たしてきたと考えられており、進化学的にも生態学的にも重要な微生物です。さらに、コケ植物から種子植物まで多くの陸上植物と共生関係を結び、作物の生育を促進することから、将来的にリン肥料の消費を押さえる微生物土壌改良材
注4としての活用も期待されています。
【研究の結果】
今回、基礎生物学研究所の前田太郎研究員、重信秀治特任准教授、川口正代司教授らを中心とした研究グループは、長鎖型次世代シーケンサー
注5を用いることで、代表的なAM菌である
Rhizophagus irregularis(図2)のゲノムを従来よりも格段に高精度に解読することに成功しました。解読されたゲノムからは、植物との共生に関わると考えられる情報伝達に関わる酵素
注6の遺伝子がAM菌で大幅に増えている一方で、脂肪酸やビタミンB1などの栄養素の生合成に関わる遺伝子を欠損しており、それらの必須栄養素を100%植物に依存している共生様式が見えてきました。従来はAM菌は植物と共生させた状態でしか培養することができませんでしたが、今後これらの栄養素を人為的に投与することによってAM菌を単独で大量培養できる可能性があります。
さらに、AM菌の種
類の同定や、植物への効果の評価に使われてきた遺伝子マーカーであるリボソームDNA遺伝子が、普通の生物では見られない特殊な性質をもつことを明らかにしました。リボソームDNA遺伝子は、形態からの識別が難しいAM菌の種を同定したり、土壌中にどのようなAM菌種がどのような割合で存在するかを調べる菌叢構造解析によく使われる重要なマーカー遺伝子です。従来の解析は、AM菌の1菌体には1つのタイプのリボソームDNA遺伝子しかないことを前提としていました。これは生物学の常識として、1個体には1つのタイプのリボソームDNA遺伝子しかないと考えられていたからです。しかし、今回のゲノム解読によって、AM菌は例外的に1菌体が10タイプのリボソームDNA遺伝子をもつことを発見しました。このような1個体に多様なリボソームDNA遺伝子を持つ生物はマラリア原虫などごくわずかしか存在しません。従来から、AM菌のリボソームDNA遺伝子がマーカー遺伝子として不可解な挙動を示すことが報告されていましたが、今回の発見により、この原因の1つがゲノムレベルでの配列の多様化にあることが明らかになりました。今後、この知見をもとにAM菌の種同定法や、植物への接種効果の評価法を改善することで、フィールドでのAM菌の種構成をより正確に評価できるようになると期待されます(図3)。
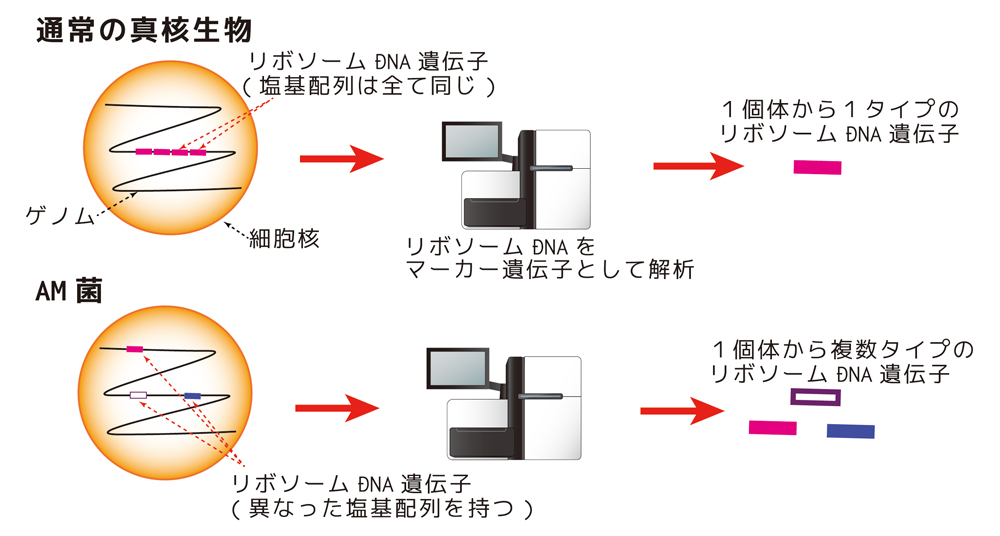 図3 AM菌リボソームDNA
図3 AM菌リボソームDNA遺伝子
のマーカーとしての特徴
通常の真核生物は、複数個のリボソームDNA遺伝子を持つが、塩基配列は全て同じであるため、シーケンサー等で解析をすると、1個体につき1つの遺伝子型が検出される。しかし、AM菌では、ゲノム内のリボソームDNA遺伝子間で配列に違いがあるため1個体から複数の遺伝子型が検出されてしまい、従来の1個体=1遺伝子型を前提としている多様性解析と齟齬をきたすことがわかった。(シーケンサーの図は© 2016 DBCLS TogoTV / CC-BY-4.0より改変)
また、1菌内に複数型のリボソームDNA遺伝子が存在することは、細胞生物学的にも興味深い事柄です。リボソームDNA遺伝子はタンパク質の製造工場であるリボソーム
注7の重要部品の設計図です。本研究によって、AM菌は1菌内に10タイプのリボソームを発現し翻訳を行っていることが示唆されました。将来的に、このような例外的な特徴がAM菌でのタンパク質生産に与える影響を調べることで、AM菌が多様な環境で生育でき、様々な植物種と共生できる仕組みが明らかになるかもしれません(図4)。
この他にも本研究グループのゲノム解読からはAM菌のリボソームDNA遺伝子が、他の真核生物と異なり直列反復構造を形成せず、これによりコピー間の多型が蓄積することで複数タイプのリボソームDNA遺伝子をもつようになったと考えられることなどが示され、今後のAM菌利用の基礎となる多くの情報を得ることができました。
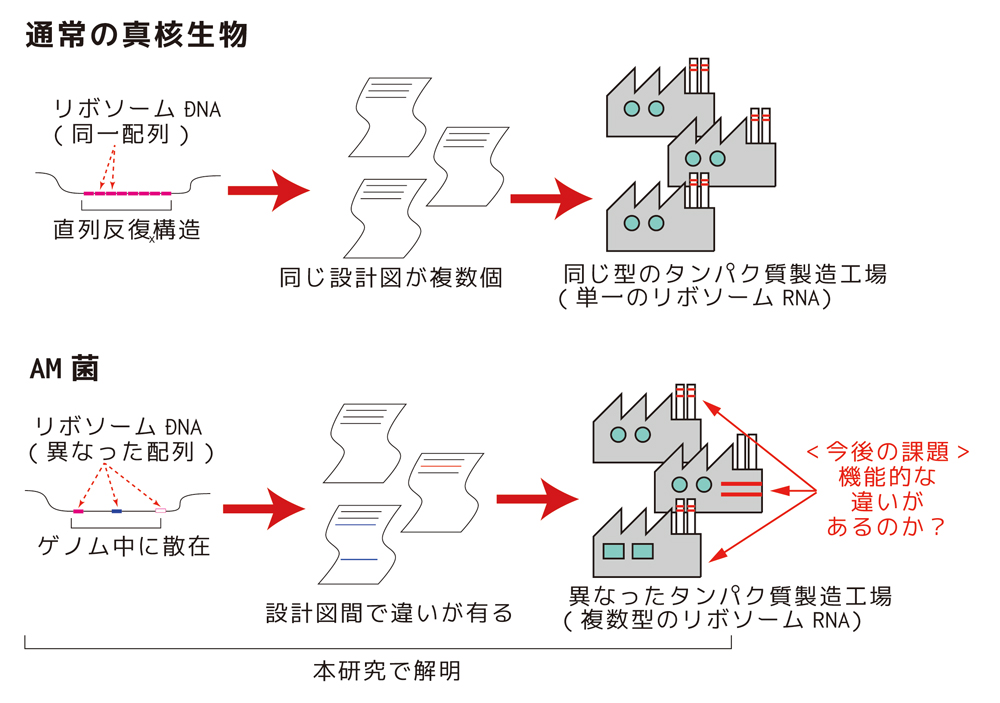 図4 まとめと展望
図4 まとめと展望
AM菌ではリボソームDNA遺伝子の散在化によりコピー間の多型が蓄積し、それは1菌内で異なった型のリボソームRNAおよびリボソームを生み出していることが示唆された。
【今後の展望】
リン肥料の原料であるリン鉱石は有限な資源であり、世界的にその有効利用が求められています。AM菌と植物の共生現象の活用は、リン肥料節減の有望な手法であり、農業を変える可能性があります。一方で、この共生現象は未解明な部分も多く、特にフィールド環境での挙動や、共生状態での栄養やシグナルのやり取りの解明にはいまだ多くの研究が必要です。
今回のゲノム解読により、AM菌の基本的なゲノム構造と遺伝子レパートリーが明確になり、AM菌の遺伝学的研究の基盤情報を整備することができました。またマーカー遺伝子における多型の発見は、従来からフィールド研究を悩ませてきたAM菌でのマーカー遺伝子の特殊な挙動の一部を説明するものです。従来のAM菌研究の障壁として、胞子を形成するには宿主との共生が必須で、単独培養ができないことがありました。植物との共存培養には手間がかかるため、播種資材が高価となり、農地での利用価値を減少させていました。また、育種や生理学的研究にも、単独培養が行えないことは障壁となっています。単独培養には、植物から与えられる栄養素やシグナル分子を補うことが必要と考えられ、今回明らかになった欠損代謝系を基にした培養実験から、単独培養が可能になるかもしれません。
【用語解説】
注1)アーバスキュラー菌根菌
植物の根に菌糸を介して共生し、皮層細胞内に栄養交換器官であるアーバスキュル(樹枝状体)を形成する菌類の総称。コケ植物から種子植物まで多様な陸上植物と共生することができる。
注2)遺伝子マーカー
ある生物個体に特有のDNA配列を言う。形態や性質で個体を識別することが難しい場合によく使われる。
注3)リボソームDNA遺伝子
リボソームの重要部品であるリボソームRNAの設計図として働く遺伝子のこと。通常生物のゲノム中には数十から数千個のリボソームDNA遺伝子が並んで存在している(直列反復構造)。
注4) 土壌改良剤
土の酸性・アルカリ性の度合いや保水性や微生物の量・活性などを変化させ栽培植物に適した環境に変えるもののことで、一般的なものに苦土石灰などがある。
注5)長鎖型次世代シーケンサー
DNAの塩基配列を読み取る機器をDNAシーケンサーと呼び、様々な読み取り技術が開発されているが、中でも次世代シーケンサーは配列解読を高度に(数千〜数億処理)並列化することで塩基の読み取りを爆発的に効率化させた機器である。従来の次世代シーケンサーは読み取り可能な配列の長さが数十〜数百塩基対であるのに対し、近年開発された長鎖型次世代シーケンサーは数千〜数万塩基対もの長いDNA断片を解析することが可能である。今回の研究では、基礎生物学研究所生物機能解析センターが運用するPacBio RSII (パシフィックバイオサイエンス社製)を利用した。
注6)酵素
化学反応を促進する生物由来の触媒のこと。多くはアミノ酸が特定の形で並んだタンパク質からなり、生命活動に必要な様々な化学変化を担っている。身近なものに消化酵素などがあり、食べた物を吸収しやすい形に変化させる。
注7)リボソーム
遺伝子情報をもとにアミノ酸をつなぎ合わせ適切な酵素活性をもつタンパク質を合成する細胞内小器官。リボソームRNAとタンパク質などから形成される。
【発表雑誌】
雑誌名: Communications Biology
掲載日: 2018年7月10日
論文タイトル: Evidence of non-tandemly repeated rDNAs and their intragenomic heterogeneity in
Rhizophagus irregularis
著者: Taro Maeda, Yuuki Kobayashi, Hiromu Kameoka, Nao Okuma, Naoya Takeda, Katsushi Yamaguchi, Takahiro Bino, Shuji Shigenobu, Masayoshi Kawaguchi
DOI:
10.1038/s42003-018-0094-7
【研究グループ】
本研究は、基礎生物学研究所の前田太郎研究員、小林裕樹研究員、亀岡啓研究員(現:大阪府立大学 JSPS特別研究員)、大熊直生研究員(現:総合研究大学院大学 博士課程)、山口勝司技術職員、尾納隆大技術職員、重信秀治特任准教授、川口正代司教授、関西学院大学の武田直也准教授による研究グループの成果です。
【研究サポート】
本研究は、JST 戦略的創造研究推進事業 ACCEL 研究開発課題名:「共生ネットワークの分子基盤とその応用展開(JPMJAC1403)」、研究代表者:川口正代司(基礎生物学研究所 共生システム研究部門 教授)、プログラムマネージャー:齋藤雅典(科学技術振興機構)の一環として実施されました。
【本研究に関するお問い合わせ先】
基礎生物学研究所 共生システム研究部門
教授 川口 正代司 (カワグチ マサヨシ)
TEL: 0564-55-7564
E-mail: masayosi@nibb.ac.jp
基礎生物学研究所 生物機能情報分析室
特任准教授 重信 秀治 (シゲノブ シュウジ)
TEL: 0564-55-7670
E-mail: shige@nibb.ac.jp
【JST事業に関すること】
科学技術振興機構 戦略研究推進部
寺下 大地
TEL: 03-6380-9130 FAX: 03-3222-2066
E-mail: suishinf@jst.go.jp
【報道担当】
基礎生物学研究所 広報室
TEL: 0564-55-7628
FAX: 0564-55-7597
E-mail: press@nibb.ac.jp
科学技術振興機構 広報課
TEL: 03-5214-8404 FAX: 03-5214-8432
E-mail: jstkoho@jst.go.jp
【研究の成果について、研究者によるより専門的な解説】
我々は、難読ゲノムの解読に効果的な「長鎖型シーケンサー」を用いることで、高精度のAM菌ゲノムの解読に成功しました。対象としたのは、モデルAM菌株として世界的に広く利用されている
Rhizophagus irregularis DAOM 181602株です。ゲノム情報は日本DNAデータバンク(DDBJ)上で公開しました(公開ID:BDIQ00000000.1)。我々のゲノム解読により、従来の国際プロジェクトでは未解明だった約800-5000万塩基を解明し、約1千〜3万箇所について新たにゲノム構造を構築し、最終的に210本の連続したゲノム配列情報(1億4975万塩基)を得ることができました。これは、予想されるゲノムサイズの97%に相当し最も正確かつ網羅的なAM菌のゲノム情報といえます。我々のゲノムからは、27,859個の遺伝子が検出され、本生物の分子生物学的解析が容易になりました。得られたゲノムデータは将来のゲノム編集技術などを使ったAM菌の遺伝子レベルの研究や育種にも利用可能と考えられます。我々のゲノムデータの解析から、脂肪酸やビタミンB1類など、他の真菌類で保存されている栄養代謝経路が複数欠損していることが明確となりました。これらの栄養素は生命活動に必須であることから、共生条件下では宿主植物から供給されていると考えられます。これらのことは従来の生理学的実験や断片的なゲノム解読からも示唆されていましたが、我々の高精度ゲノムでも検証されたことで、これらの栄養素が遺伝子レベルで合成できないことが明確となり、宿主との栄養のやり取りの重要性が明確になりました。
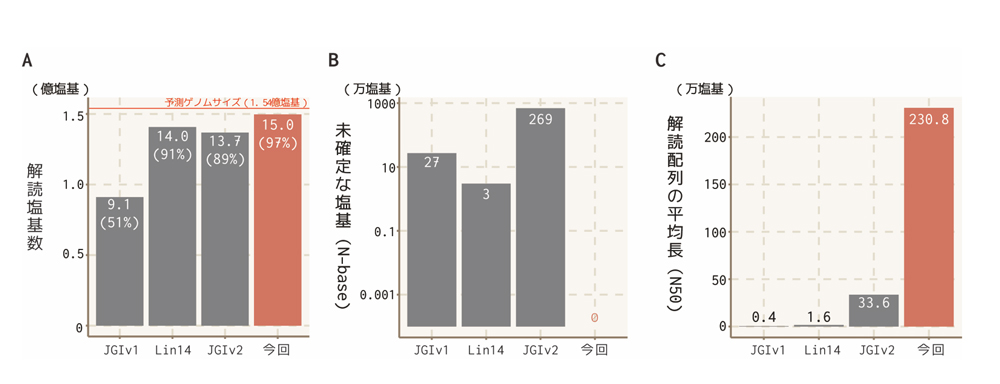 補足図1:構築されたゲノム品質を先行研究の結果と比較
補足図1:構築されたゲノム品質を先行研究の結果と比較
我々のゲノムは先行研究より幅広いゲノム領域を解明した(A)。先行ゲノムでの解読領域には、未確定な塩基部位が数万以上含まれていたが、我々のゲノムでは未確定領域は存在しない(B)。従来の配列情報は断片化が激しかったが、我々はゲノムデータ断片化の基準となる加重平均配列長(N50)が100万塩基を上回る高品質のデータを得た。JGIv1; Tisserant
et al. (2013)
Proc Natl Acad Sci U S A., Lin14; Lin
et al. (2014)
PLoS Genet., JGIv2; Chen
et al. (2018)
New Phytologist.
従来の断片化ゲノムでは解析が困難だったリボソーマルDNAについて、ゲノム中のすべての遺伝子コピー(相同遺伝子)を解読し、それらの位置関係を明らかにしました。結果、これらのリボソーマルDNA遺伝子間には100塩基あたり約4塩基と非常に多くの多型部位があることがわかりました。リボソーマルDNAは、生態学や系統学のマーカー遺伝子として頻用されており、AM菌でも例外ではありません。しかし、その解析法はいくつかの前提に基づいており、その一つはゲノム上に複数あるリボソーマルDNA遺伝子の配列が全て同一であり、1個体につき1マーカー型を持つとみなせるということです。今回、AM菌ではこの前提が通用せず、1個体が複数マーカー型をもつことが明らかになりました。そのため、従来のAM菌の多様性解析は土壌中のAM菌の多様性を過大評価していたり、播種菌株を正確に追跡できていないなどの可能性があり、再検討の必要があると考えられます。
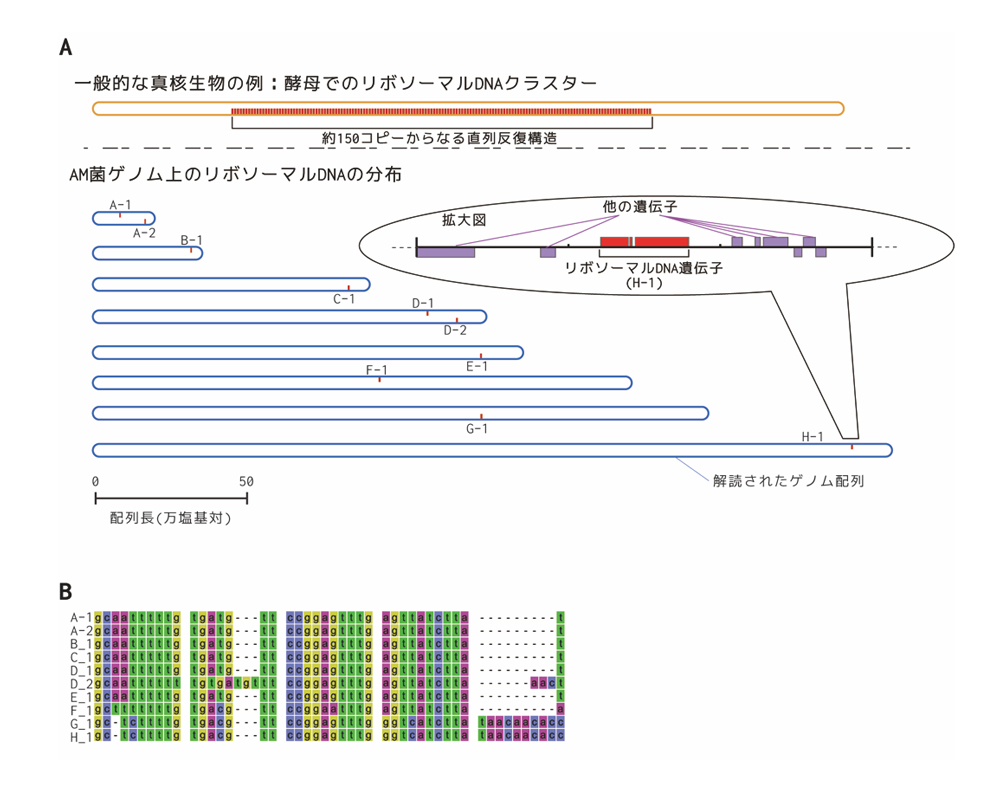 補足図2:AM菌リボソームDNAのゲノム内多型と分布
補足図2:AM菌リボソームDNAのゲノム内多型と分布
我々のゲノム解読で判明したAM菌のリボソーマルDNAのゲノム内の位置関係(A)とその配列レベルでの違い(B) A) 酵母などほとんどの真核生物ではリボソーマルDNAはゲノム上の特定の位置に直列反復構造をとって分布しているが、AM菌ではこの構造は見られず、ゲノム上に10個の配列として散在している(コピー数はRead depthにより確認)。B) AM菌のリボソーマルDNAをアライメントした結果(一部)。コピー間で顕著な配列の違いがあることがわかる。
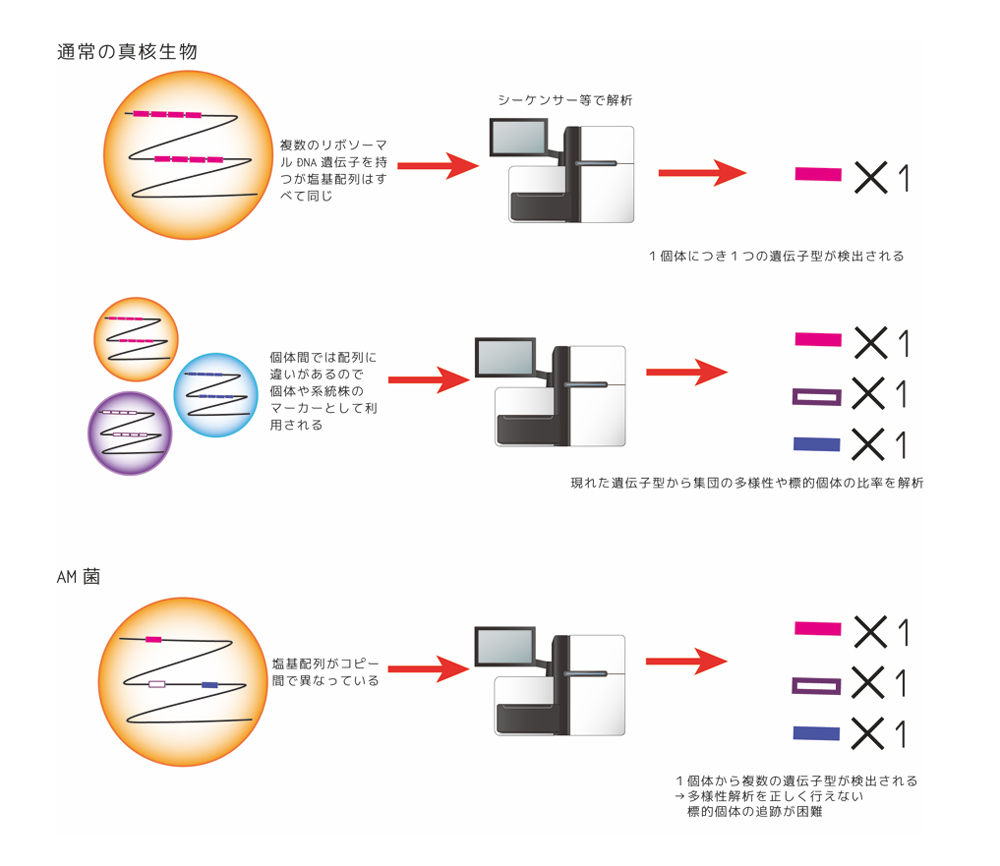 補足図3:AM菌リボソームDNAのマーカーとしての特徴
補足図3:AM菌リボソームDNAのマーカーとしての特徴
AM菌では、ゲノム内多型があるため1個体から複数のリボソーマルDNAの遺伝子型が検出されてしまい、従来の1個体から1遺伝子型を前提としている多様性解析と齟齬をきたす。(シーケンサーの図は© 2016 DBCLS TogoTV / CC-BY-4.0より改変)
今回多型が見つかったリボソーマルDNAは生物がつくる全てのタンパク質の製造工場であるリボソームの重要構成因子リボソーマルRNAをコードしており、ここに多型があることは真核生物として非常に例外的です。リボソーマルRNAに着目したTotal RNAseq 法により、我々はすべての多型リボソーマルDNA遺伝子が、リボソーマルRNAを作ることができ、多型はリボソーマルRNAになっても維持されることを確認しました。現在我々は、AM菌が機能が異なった複数型のリボソームの使い分けによって表現型を変化させている可能性に注目しています。将来的に、この多型リボソーマルDNAの役割の解明によって、AM菌の個体間での表現型可塑性(同じ親胞子由来の株でも植物への効果が異なる等)の解明や、幅広い植物種に共生できる宿主可塑性の機構解明、そしてより安定した農業的有効株の選抜につながるのではないかと考えています。
我々のゲノム解読は、リボソーマルDNA遺伝子同士の位置関係も明らかにすることができました。そしてこの結果はリボソーマルDNAの多型が生じる進化的機構について示唆をもたらすものでした。通常の真核生物ではリボソーマルDNAはゲノム上の特定の位置に数十から数千個の直列に並んだ「直列反復構造」を作ります。この反復構造を使って、効率的に遺伝子コピー数を増やすことで、十分なコピー数を維持するとともに、コピー間の配列の違いが起きにくくなっているとされています(協調進化 Concerted evolution)。我々の結果から、AM菌ではこの直列反復構造が崩壊していることが明らかになりました。これによって、通常のリボソーマルDNAのような協調進化が働かず、ゲノム内多型の蓄積につながっていると考えられます。このタンデムリピート構造の欠損は真核生物では他にマラリア原虫でしか報告がない非常に例外的な特徴です。今後の、AM菌における直列反復構造崩壊の適応的意義に関する研究によって、真核生物におけるリボソーム遺伝子の進化過程の一部が明らかになるかもしれません。




 図1 アーバスキュラー菌根菌と植物の共生現象の概念図
図1 アーバスキュラー菌根菌と植物の共生現象の概念図 図2 アーバスキュラー菌根菌と植物根
図2 アーバスキュラー菌根菌と植物根 図3 AM菌リボソームDNA遺伝子のマーカーとしての特徴
図3 AM菌リボソームDNA遺伝子のマーカーとしての特徴 図4 まとめと展望
図4 まとめと展望 補足図1:構築されたゲノム品質を先行研究の結果と比較
補足図1:構築されたゲノム品質を先行研究の結果と比較 補足図2:AM菌リボソームDNAのゲノム内多型と分布
補足図2:AM菌リボソームDNAのゲノム内多型と分布 補足図3:AM菌リボソームDNAのマーカーとしての特徴
補足図3:AM菌リボソームDNAのマーカーとしての特徴