バイオインフォマティクスを専門としない生物学研究者を対象に、次世代シーケンシング(NGS)技術を使ったトランスクリプトーム解析(RNA-seq)をどのように実験デザインし、どのように膨大な遺伝子発現データから生物学的な情報を抽出するのか、その基礎的技術と考え方を身に付けることを目的としたコースです。 次世代シーケンスデータのフォーマットの理解などの基礎的事項から、ゲノム情報のない生物種でトランスクリプトーム解析を可能にする
de novo RNA-seq 解析などの発展的内容までカバーします。講義とコンピュータを用いた演習を組み合わせて行います。
本コースは、準備編と実践編の2つから構成され、それぞれ2日間の日程で開催されます。準備編では、データ解析を行う際の共通の基盤として、Unixオペレーティングシステム、および統計解析パッケージRの基本的な使い方を学習します。UnixやRのコマンドを使った基本的な処理の流れを習得することを目的としますが、演習では次世代シーケンサーのデータ解析を想定した例題も扱います。実践編では、RNA-seqによる発現変動解析の原理を理解した上で、実践的なデータ解析パイプラインを習得します。
準備編

 実践編 *オンライン開催
実践編 *オンライン開催
 受講者数
受講者数
準備編26名(応募109名)・実践編33名(応募53名)
開催報告
ゲノムインフォマティクス・トレーニングコース (GITC) は、次世代シークエンサー (NGS) の登場により、大規模なシークエンスデータを解析する必要に迫られた実験生物学者を対象としたインフォマティクス技術のトレーニングコースです。手持ちのデータを解析するために直ちに必要となるプログラムの使い方など実践的な内容に加えて、大規模なデータ解析を行う際に必要となる計算機操作や統計的な考え方など、実験生物学者があまり触れてこなかったと思われる基礎的な内容にも力を入れた構成になっています。「RNA-seq入門」は、「準備編」と「実践編」の 2 部構成となっており、「準備編」は解析プラットフォームとしてのUNIXやRの使い方とNGSデータの基本的な取扱いについて習熟することを、「実践編」は実際にRNA-seqデータを処理し、統計解析を行って生物学的に有用な結果を得るまでの流れを習得することを目標とします。通常、両者は2~3週間の間をおいて開催され、両方を通して受講することで、まったくの初心者の方でも、手持ちのデータを解析するために必要な一通りの知識と技術が身につくような構成になっています。また受講者の準備状況や習熟度によっては、どちらか一方のみに参加することも可能です。GITCの看板コースであり、近年は応募者が100名を大きく超えるようになっています。
ただ、本コースは、新型コロナウィルス感染症の拡大が懸念される中での開催となり、結果として「準備編」は一度延期して当初の「実践編」の日程で実施し、「実践編」の方は次年度に延期することとなりました。 また実施した「準備編」においても、参加者のキャンセルが相次ぎ、当日は参加者間の情報交換の場として予定していた懇親会を中止したほか、プログラム内容を一部変更し、またマスク着用など感染防止に配慮した形での開催となりました。
「実践編」はオンラインで次年度6月に開催しました。講義形式の授業とハンズオン形式のコンピュータ演習を並行して行いますが、特に初心者がつまずきやすいコンピュータ演習には、フロアサポートによるきめ細かい対応を行っています。これをオンラインでどう実現するかが問題でしたが、Zoomによる講義とチャットによる個別の質問対応を組み合わせて行うことにしました。また、コンピュータ実習の主要部分は基生研のサーバにログインして行うことで、受講生の準備の負担を減らすようにしました。当日は不安もありましたが、幸い大きなトラブルもなく、懸案となっていたチャットでの質問対応を含めて、受講生からの評価は概ね良好でした。
オーガナイザー 内山 郁夫(生物機能解析センター 情報管理解析室)

Program
「準備編 : UNIX・R・NGSの基本」 2020年2月27日(木) 10:00~ 28日(金)17:30
UNIX基礎
シェルスクリプト
R入門
NGS基本データフォーマット
NGS基本ツール
テキスト処理
統計学入門
演習
「実践編 : RNA-seq解析パイプライン」 2020年6月4日(木) 10:00~ 5日(金)17:30 *オンライン開催
RNA-seq入門 概論
NGS基本データフォーマット復習
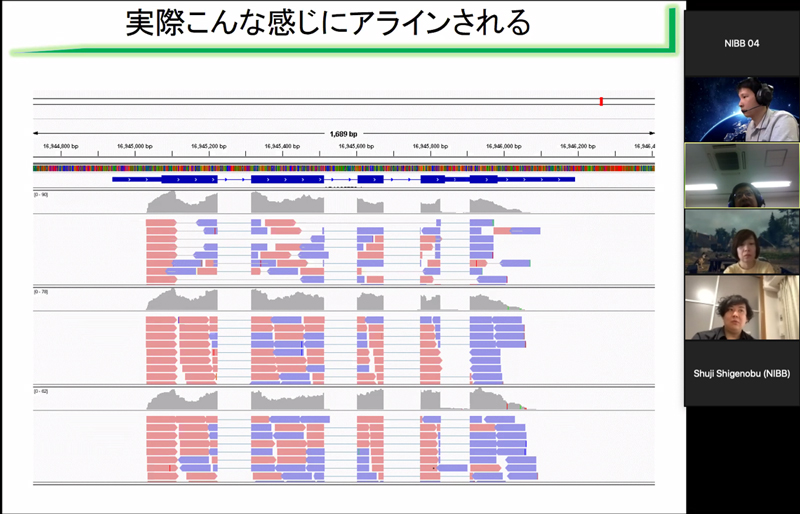
NGS基本ツール:Bowtie2、samtools、IGVなど
RNA-seq基礎・トランスクリプトベース・ゲノムベース・de novo
多変量解析
機能アノテーションと GO 解析
実践演習
まとめ









