バイオインフォマティクスを専門としない生物学研究者で、これから次世代シーケンシング(NGS)データの解析を行おうとしている方を対象に、ゲノムインフォマティクスの基礎的技術と考え方を身に着けることを目的としたコースです。
本コースは2日間の日程で開催されます。RNA-seq入門では、RNA-seqによる発現変動解析の原理を理解した上で、実践的なデータ解析パイプラインを習得します。
 受講者数
受講者数
受講生 27 名 聴講生 12 名 (応募総数 187 名)
開催報告
ゲノムインフォマティクス・トレーニングコース (GITC) は、次世代シークエンサー (NGS) の登場により、大規模なシークエンスデータを解析する必要に迫られた実験生物学者を対象としたインフォマティクス技術のトレーニングコースです。手持ちのデータ解析に直ちに必要となるプログラムの使い方など実践的な内容に加えて、大規模なデータ解析を行う際に必要となる計算機操作や統計的な考え方など、実験生物学者があまり触れてこなかった基礎的な内容にも力を入れた構成になっています。GITCには複数のコースがありますが、いずれも計算機によるハンズオン実習を含んでおり、受講者個々のスキルに応じてきめ細かいサポートを行う点が特徴となっています。「NGS解析入門」は、解析プラットフォームとしてのUNIX やRの使い方とNGSデータの基本的な取扱い、統計的な考え方などを習得する基礎的なコース、「RNA-seq入門」は、実際に RNA-seqデータを処理し、統計解析を行って生物学的に有用な結果を得るまでの流れを習得する実践的なコースで、両者を合わせて受講することで、RNA-seq解析の基礎から実践までを学ぶことができます。
今年度はNGS解析入門とRNA-seq入門を各2回、いずれもオンラインで開催しました。オンライン開催によって、来所が難しい方も参加いただけるようになり、いずれも両コースを合わせて200名を超える応募がありました。残念ながら、きめ細かいサポートを行うために、受講生の数は30名弱程度に絞らざるを得ませんでしたが、昨年度同様にサポートを限った「聴講生」の参加枠を設けることで、少しでも多くの方に参加いただけるようにしました。
オンライン開催も2年目に入ってスムーズに進むようになりました。実習は、基本的にあらかじめ実習環境が整備された共通計算機にログインして行う形にしましたが、併せて受講生が自身で環境構築を行う際のマニュアルも整備し、グラフィカルな表示を行うRなどは、ローカルで実行するようにしました。また、利用者の個別の質問への対応はSlackを用いて行いましたが、他人の質問も含めて回答が随時閲覧できることから、以前より便利になったと概ね好評でした。一方、参加者間の交流が難しいことがオンライン開催の欠点のひとつですが、それを補うために休憩時間などを利用して、自己紹介や雑談の時間を設けるなどの工夫もしました。
オンライン開催が好評であることから、今後コロナ禍が収束したとしても、何らかの形でオンライン開催の有利な点は継続していくことになると思われます。今後とも内容や方式についての検討を重ね、より良いコースにしていきたいと思います。
オーガナイザー 内山 郁夫(生物機能解析センター 情報管理解析室)

Program
「RNA-seq入門 : RNA-seq解析パイプライン」 2022年3月2日(水)9:30~ 3日(木)18:00
RNA-seq入門 概論
NGS基本データフォーマット復習
NGS基本ツール:Bowtie2、samtools、IGVなど
RNA-seq基礎・トランスクリプトベース・ゲノムベース・de novo
多変量解析
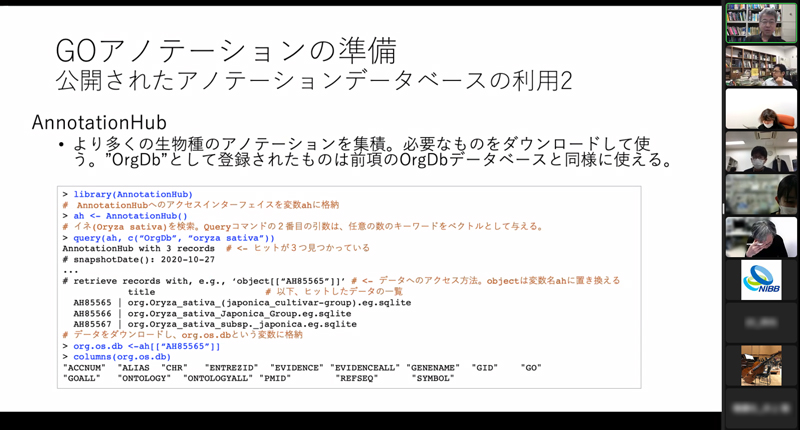
機能アノテーションと GO 解析
実践演習
まとめ







