バイオインフォマティクスを専門としない生物学研究者を対象に、次世代シーケンシング(NGS)技術を使ったトランスクリプトーム解析(RNA-seq)をどのように実験デザインし、どのように膨大な遺伝子発現データから生物学的な情報を抽出するのか、その基礎的技術と考え方を身に付けることを目的としたコースです。
本コースは2日間の日程で開催されます。RNA-seq入門では、RNA-seqによる発現変動解析の原理を理解した上で、実践的なデータ解析パイプラインを習得します。
 受講者数
受講者数
受講生 30 名 聴講生 27 名(応募総数 100 名)
開催報告
ゲノムインフォマティクス・トレーニングコース (GITC)は、次世代シークエンサー (NGS) の登場により、大規模なシークエンスデータを解析する必要に迫られた実験生物学者を対象としたインフォマティクス技術のトレーニングコースです。手持ちのデータ解析に直ちに必要となるプログラムの使い方など実践的な内容に加えて、大規模なデータ解析を行う際に必要となる計算機操作や統計的な考え方など、実験生物学者があまり触れてこなかった基礎的な内容にも力を入れた構成になっています。GITC には複数のコースがありますが、いずれも計算機によるハンズオン実習を含んでおり、受講者個々のスキルに応じてきめ細かいサポートを行う点が特徴となっています。「NGS 解析入門」は、解析プラットフォームとしての UNIX や R の使い方と NGS データの基本的な取扱い、統計的な考え方などを習得する基礎的なコース、「RNA-seq 入門」は、実際に RNA-seq データを処理し、統計解析を行って生物学的に有用な結果を得るまでの流れを習得する実践的なコースで、両者を合わせて受講することで、 RNA-seq 解析の基礎から実践までを学ぶことができます。
今年度はRNA-seq入門のみのコースを夏に、 NGS 解析入門と RNA-seq 入門のフルコースを冬に開催しました。夏のコースは昨年度と同様にオンラインのみで開催しましたが、コロナ禍が収束してきた冬のコースでは、GITCでははじめてオンサイト、オンラインのハイブリッドで開催しました。ハイブリッド開催では、コロナ前に行っていたオンサイトでの受講生へのきめ細かい対応と、遠隔地からでも気楽に参加できるというオンラインのメリットを両立させるように配慮しました。その結果、オンラインの参加者は聴講のみで個別の対応は一切しないという条件ながら、希望者はほぼ全員が参加できるようにしました。一方で、コース全体に参加して一定の課題をこなした受講生に対しては、今年度からコース修了証を発行することにしました。
コロナ禍がようやく収束し、オンサイトで顔をつきあわせての集会も可能になってきましたが、一方でここ数年でオンライン開催の便利さが浸透してオンラインでの参加を希望される方も増えています。オンライン、オンサイトのそれぞれのメリットを生かした開催方法を含めて、今後とも内容や方式についての検討を重ね、より良いコースにしていきたいと思います。
オーガナイザー 内山 郁夫(超階層生物学センター データ統合解析室)

Program
「RNA-seq入門 : RNA-seq解析パイプライン」 2022年8月31日(水)10:00~ 9月1日(木)18:30
RNA-seq入門 概論
NGS基本データフォーマット復習
NGS基本ツール:Bowtie2、samtools、IGVなど
RNA-seq基礎・トランスクリプトベース・ゲノムベース・de novo
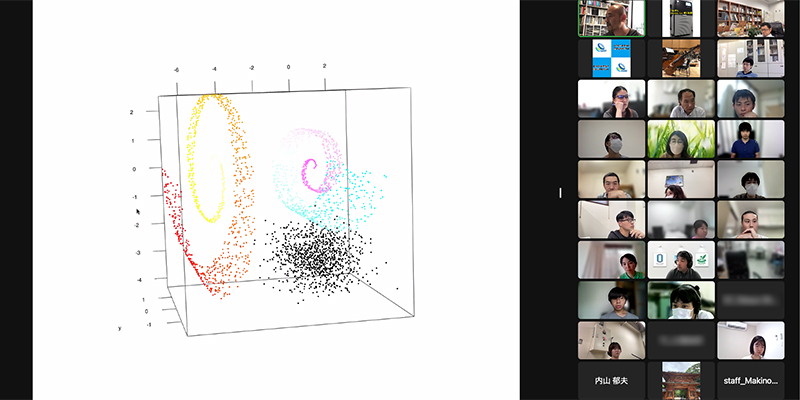
多変量解析
機能アノテーションと GO 解析
実践演習
まとめ







