
基礎生物学研究所




私たちの個体を形作る細胞は、それぞれ全く同じセットのゲノムDNAを持っています。しかし、個々の細胞の働きは多様であり、全ての細胞が同じようにDNAを使っていたのでは、このような多様性を生み出すことはできません。このように、DNAの一次配列だけでは説明できない現象を対象とする研究分野は「エピジェネティクス」と呼ばれ、個体の発生や細胞の分化だけでなく、疾患や老化のメカニズム解明の鍵を握る研究と考えられています。私たちはDNAを取り巻く「クロマチン」という構造に着目し、そのダイナミックな構造変換がどのようにエピジェネティックな現象を調節しているのか、その分子機構の解明を目指しています。
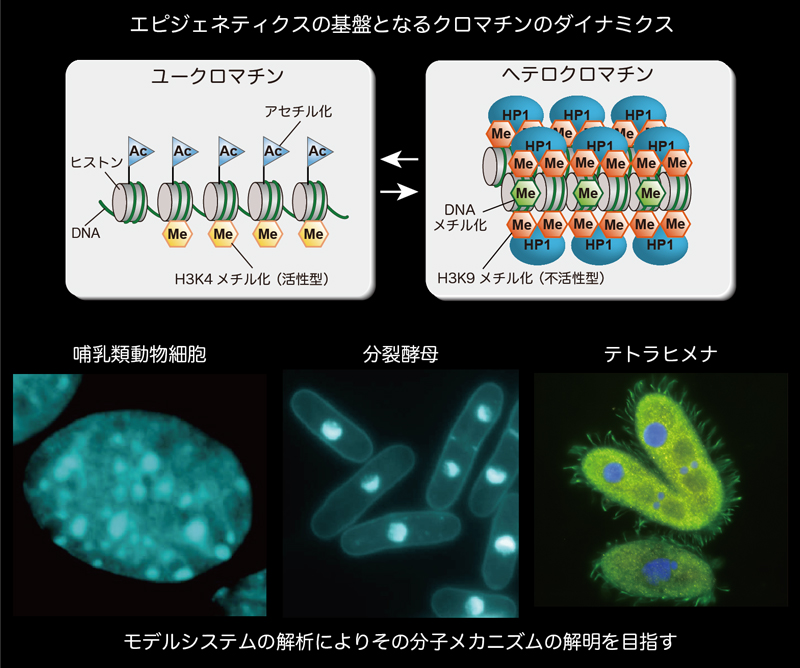
(上段)ユークロマチン領域(左)では、転写の活性化に関わるヒストンのアセチル化や、H3K4メチル化が存在している。一方ヘテロクロマチン領域(右)では、抑制的に働くH3K9メチル化やDNAメチル化が存在し、進化的に保存されたHP1タンパク質が結合して高次のクロマチン構造が形成される。
(下段)当研究部門では、哺乳類動物細胞、分裂酵母、原生生物テトラヒメナなどを用いて、クロマチンのダイナミックな構造変化をもたらす分子機構の解明を目指している。
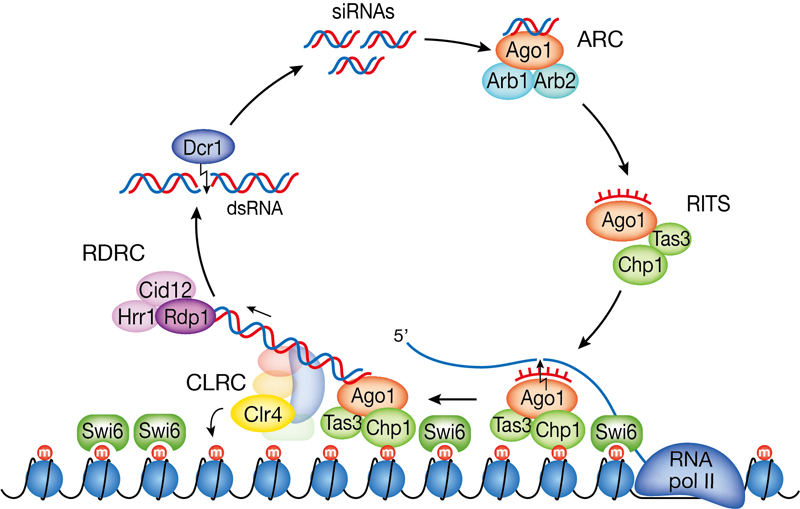
真核細胞の染色体には、高度に凝縮したヘテロクロマチンと呼ばれる構造が存在している。この高次のクロマチン構造は、セントロメアなど、染色体の機能ドメインの形成に寄与するとともに、エピジェネティックな遺伝子発現の制御にも重要な役割を果たしている。分裂酵母などのモデル生物を用いた解析から、ヘテロクロマチンの形成に、RNAサイレンシングと呼ばれる機構の関与が明らかにされた。しかし、高次クロマチン構造がどのように形成され維持されているのか、その分子機構にはまだまだ数多くの謎が残されている。私達の研究部門では、種々のモデル生物を用いてヘテロクロマチン構造の形成メカニズムの解明に取り組んでいる。
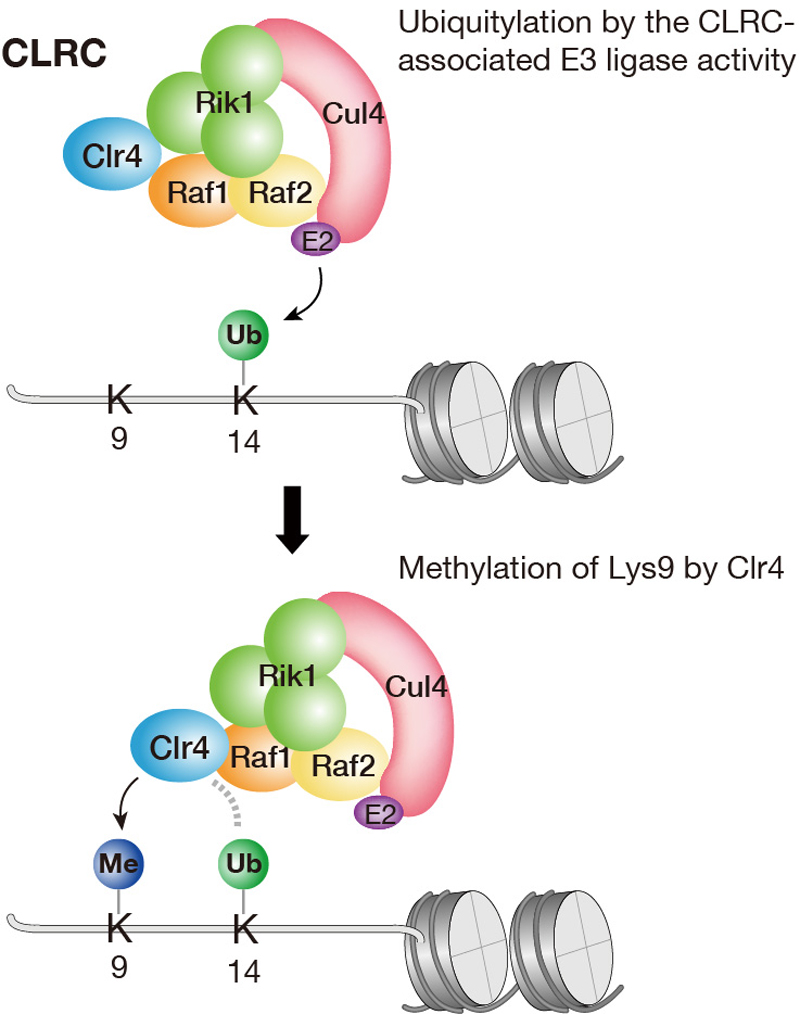
クロマチンの大きな構造変化や、遺伝子発現を調節するためには、クロマチンの基本単位であるヌクレオソームの構造を変化させる必要がある。近年の解析から、ヌクレオソームを構成するヒストンが多様な翻訳後修飾を受け、この変化が様々な生命現象と関わることが明らかにされてきた。特にヒストンのメチル化修飾は、安定なエピジェネティックマークと考えられており、そのメチル化修飾の変化を制御している機構の解明は、エピジェネティックな遺伝子発現制御の理解につながると期待されている。私達の研究部門では、ヒストン修飾酵素の制御機構の解明を目指している。
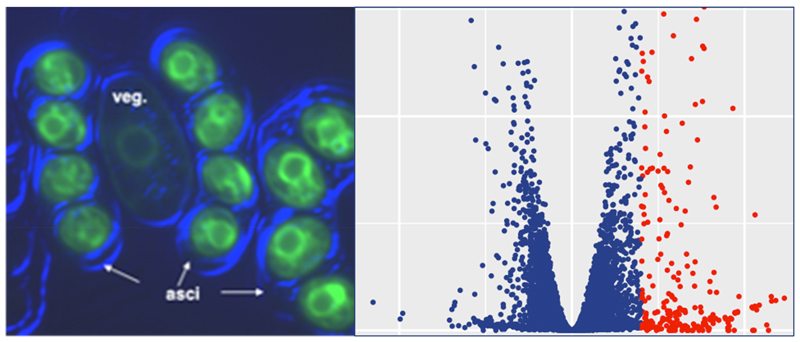
有性生殖を行う生物は、減数分裂の過程を経て半数体である配偶子や胞子を形成する。配偶子や胞子は、ゲノムDNAの情報に加えて遺伝子発現の記憶に相当するエピジェネティックな情報も伝播していると考えられるが、それらの情報がどのように特殊なクロマチン構造を介して伝播されるのか、その分子機構については不明な点が多く残されている。私たちの研究部門では、高等真核生物の配偶子のモデルとして分裂酵母の胞子を研究材料に用いて、エピジェネティックな情報が世代を超えて伝播される仕組みの解明を目指している。
私たちの身の回りには、DNAだけでは説明できない現象がたくさんあります。あなた自身もDNA以上の物を両親から受け継いだ存在です。私たちの研究部門では、そのような生命の不思議に興味を持ち、私たちと一緒に分子のレベルで研究してみたい、という学生を募集しています。研究室の見学も大歓迎です。
中山 潤一 教授 E-mail: jnakayam@nibb.ac.jp
