
基礎生物学研究所





システム生物学的アプローチによる細胞モデリングでは、生体分子情報を基にして転写、代謝、シグナル伝達などの生命機能を数理モデル化し、複雑な細胞をシステムとして理解するための基盤を築いてきました。例えば、大腸菌やマイコプラズマなどの全細胞モデリングでは、多種多様な生体分子情報を反応速度式に変換してゲノム配列から表現型を予測し実証しました(Ahn-Horst et al., NPJ Syst. Biol. Appl. 2022; Karr et al., Cell 2012; Tomita et al., Bioinform. 1999)。しかし、細胞は単なる生物分子ネットワークの集合体ではなく、熱学・光学・流体力学・統計力学・量子生物学などから導き出された支配方程式と多様な物理パラメータも同時に内包しており、生体分子のネットワーク情報と相関を持つことで一つの生命システムとして成り立っています。本研究室では、細胞内で成り立つべき物理法則や支配方程式(熱伝導方程式や波動方程式など)に基づき、生命情報と物理情報を同時に取り扱うことのできる新しい細胞モデリングの手法を開発しています。
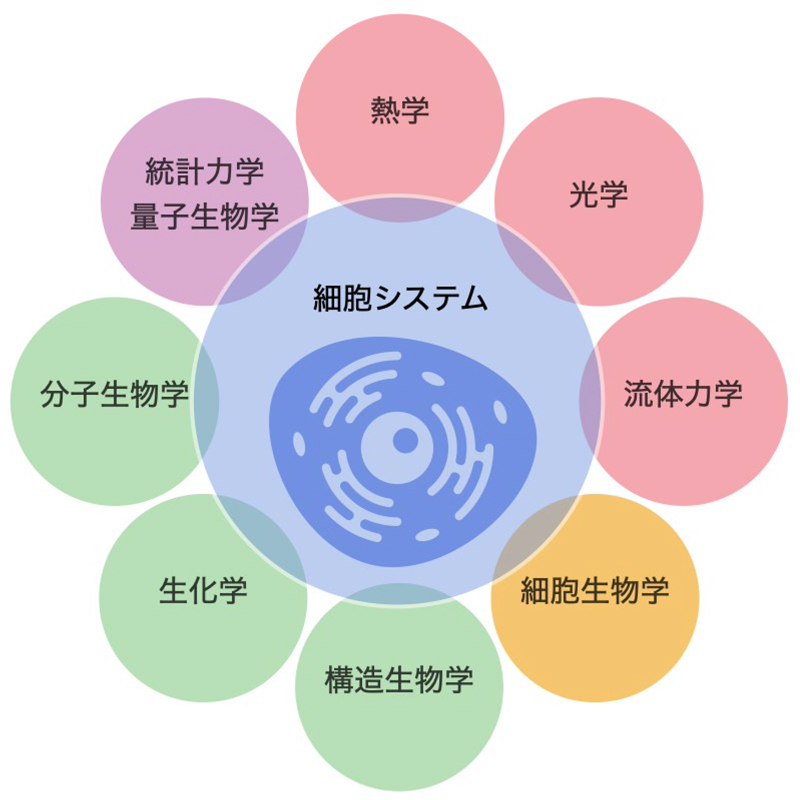
システム生物学に基づいた細胞モデリングでは、生物学的なパラメータと分子反応ネットワークが主に注目されてきたが、我々は細胞という一つのシステムを現象論的視点から探究し、細胞内のミトコンドリア熱源の分布・温度分布・熱容量・熱伝導率・屈折率・散乱係数・粘性係数・流体の速度場などのマクロな物理情報も含めたより広範な視野で理解しようと試みている。
これまでの研究活動では、特に、光が細胞内を伝播するプロセスに焦点を当てて細胞モデリングを行なってきた。2022年の研究では、細胞内の屈折率乱流を表す光学フラクタルモデルを用いて、光学系における波動関数の「振幅」と「位相」の数学的相関を表す強度輸送方程式(TIE)の再公式化を行った(文献1)。広範囲の波長領域においてTIEを数値シミュレーションすることで、強度分布がどのようにしてフラクタル構造と相互作用して分散・減衰するのかを明らかにした(図1)。また、細胞内の位相分布とWhittle-Matern光学相関を数学的に結ぶ関数を導出することに成功し、蛍光画像から細胞内の屈折率ゆらぎ・フラクタル次元・散乱係数などの光学的特性を再構築することが可能になった。

図1. 標準画像”Pepper”からなる平面波(波長507nm)が、TIEに従って細胞内の光学フラクタル構造を伝播するプロセスをシミュレーション実装した結果。
細胞モデルが予測する生命現象(または、細胞モデルに組み込まれた仮説)を実験的に検証するためのツールとして「生物画像シミュレーション」の実装にも取り組んでいる。これまでの研究活動では、蛍光イメージングによる画像生成の光学プロセスをシミュレーション実装するための基盤を提供し、蛍光顕微鏡(TIRFM)やレーザー走査型共焦点顕微鏡(LSCM)などのシミュレーション・モデュールの実装をしてきた。特に、2019年の研究では、遮光照明顕微鏡シミュレータを用いて一分子蛍光画像を生成し、実際の一分子蛍光画像とフォトンカウンティングの単位で直接比較(図2)できるようにしてきた。また、一分子計測をする際に発生する非経験的な不確実性が、どのようにして細胞膜受容体の協同結合の識別に影響し、生物学的な意味の誤解釈に繋がるかを明らかにした(文献3)。
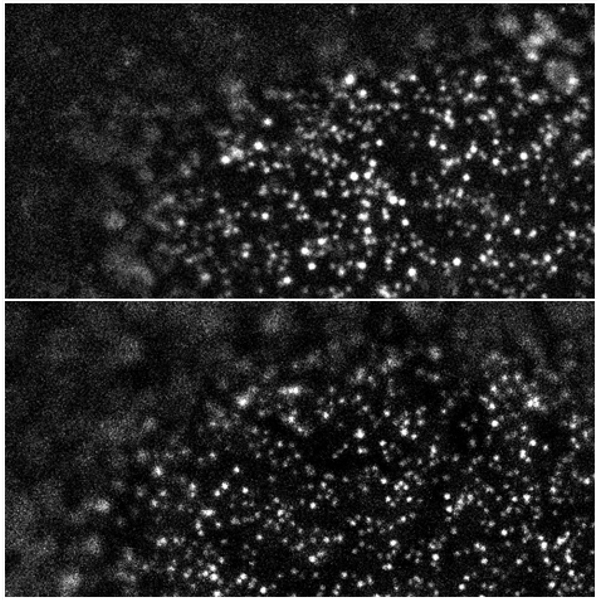
図2. 遮光照明顕微鏡による実際の一分子蛍光画像(Hiroshima et al., PNAS 2012)(上図)と遮光照明顕微鏡シミュレーションによる一分子蛍光画像(下図)。