
基礎生物学研究所





超低温の世界で生物を保存するためには細胞の適応が必要です。その条件を探索する生物遺伝資源の保存技術の開発を行なっています。超低温の世界ではDNA配列が変化しなくても、様々な変化が起きています。ゲノム解析技術の進展により、様々な生物の分子生物学的な解析が可能となってきました。その中で保存方法の確立していない生物を対象に、低温・超低温条件下における反応を生体・物質の面から解析し理解することを目指しています。また、ゲノム中には多くの転移因子(トランスポゾン)が存在していますが、その多くは転移できません。しかし稀にゲノムによる抑制をすり抜けて転移できるトランスポゾンが存在してゲノムの再編成を起こしています。どのようにゲノムはトランスポゾンを制御しているのか、また転移によって引き起こされるゲノムの再編成は生物にどんな影響を与えているのかを調べています。

茎頂や腋芽を超低温で保存することがてきれば、接木によって親個体と同じゲノムを持った植物を再生することができる。カンキツ類はゲノムのヘテロ性だけでなく、種子の多胚性のために種子で親の性質を維持することができにくい。
植物の種子は優れた保存器官であるが、寿命の短い難保存性の種子もあるので種子の寿命を伸ばす環境条件や要因について研究している。またカンキツなどは、ゲノムのヘテロ性が高いために種子では有用な形質を種子で遺伝させることができない。これらの植物は茎頂を保存することで系統の保存を行うことが必要となる。カンキツは低温に弱いので、低温に適応させた保存技術の開発を行っている。
多くの生物のゲノム中には多くのトランスポゾンが存在している。トランポゾンによるゲノムの再編成は、進化の原動力一つとなっていると考えられるが、トランスポゾンの転移は、ホストのゲノムにとって有害になるので、転移する能力はジェネティックやエピジェネティクに抑制されており、通常の成育条件下で転移する事はまれである。そこで転移できるDNAトランスポゾンに注目して、トランスポゾンによるゲノムのダイナミズムと遺伝子発現の制御機構の解明を明らかにすることを試みている。
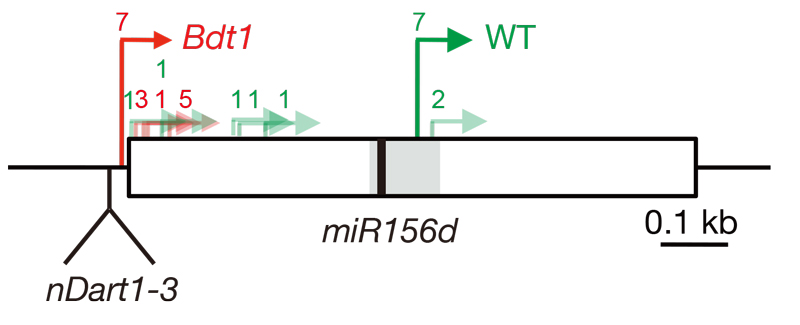
図1. nDartの挿入による優性変異の原因の解明(文献5)
我々は自然栽培条件下で活発に転移することができるDNAトランスポゾンnDart1を同定した。nDart1の転移には、自律性因子nDart1が必要であるが、通常はエピジェネテイックに抑制されている。nDart1が活発に転移する時期を明らかにし(文献2)、さらに、脱メチル化によってnDart1を持たないイネ系統でも転移を活性化できることも示した(文献4)。nDart1は、GC含量の差が大きい領域に挿入し易い性質をもっているので、ゲノム中に存在している転移の制御因子の同定に向けて研究を行っている。
ゲノムの変異の多くは劣性となるが(文献5)、nDart1の挿入変異体の中にはしばしば優性となる突然変異体が観察される。不完全優性でわい性となるBdt1変異体では機能のあるマイクロRNAの発現様式がnDart1の挿入で変化していた(図1, 文献3)。DNAトランスポゾンが優性変異の原因となる例は非常に珍しく、その原因は未解明な部分が残されているので、優性となった変異体を選抜して解析を行っている。
nDartの挿入変異体を利用した研究・植物の種子や組織の低温・超低温保存方法の開発について共同研究に応じることができます。
栂根 一夫 助教 E-mail: tsugane@nibb.ac.jp