研究の概要
Reading, Editing & Reconstructing
全ての生物のゲノム配列を解読し(Reading)、その配列を個体レベルで編集(Editing)できる時代が到来しています。これまで、モデル生物のみに用いられてきた分子生物学的・逆遺伝学的研究手法を、あらゆる生物へ適応することが可能になりつつあります。21世紀の基礎生物学は、地球上の生物が営む生命現象を広く深く探究する、新しい学問へと生まれ変わろうとしています。基礎生物学の発展に向けて、我々のグループではゲノム編集(Editing)技術や新規モデル生物の開発、および動物の組織再構築現象(Reconstructing)に関する研究を行なっています。

右が CRISPR-Cas9で色素合成遺伝子を破壊(ノックアウト)した当世代のイベリアトゲイモリで左は野生型。
CRISPR-Cas9によるゲノム編集効率が非常に高いこともイベリアトゲイモリの特徴の一つである。
Reading & Editing
次世代シークエンサー(NGS)の開発と普及により、研究者は対象生物のゲノム配列を決定し、生命現象に関わる全ての遺伝子発現の情報を得ることが可能になった。また、高性能質量分析計を用いてタンパク質や代謝物を網羅的に同定することも容易となった。もはや、生命現象を司る分子群(要素)のほぼすべてを明らかにすることができると言えるだろう。さらに、CRISPR-Casに代表される人工DNA切断酵素によってゲノム配列を操作する「ゲノム編集技術」が登場したことで、生物学は一転しようとしている。これまで、 任意の遺伝子を破壊(ノックアウト)したり、外来遺伝子を任意の場所に挿入(ノックイン)したりする技術は、モデル生物と呼ばれるごく一部の生物種に限られた逆遺伝学的ツールとして用いられてきたが、現在では理論上全ての生物への適応が可能となっている。興味を持った生物や生命現象について、生物学者が思う存分研究できる時代を迎えている。
我々のグループは、主に両生類を用いてゲノム編集技術を開発し、様々な新規モデル生物種への支援を行なっている。また、新規モデル生物であるイベリアトゲイモリを器官再生研究の中心とすべく、その研究基盤の整備にも力を入れている。我々は、CRISPR-Cas9を用いた超高効率な遺伝子ノックアウトによって、ファウンダー(当世代)での迅速な遺伝子機能解析を実現した。この研究戦略は、NGS解析により見つかる大量の遺伝子や転写調節領域を解析するうえで、大いに期待されている。また、両生類における世界初の遺伝子ノックインに成功し、その際に開発した新技術は、現在様々な生物種に応用されている。
Reconstructing
両生類が見せる再生と変態(メタモルフォーゼ)はとてもユニークな生命現象である。無尾両生類のオタマジャクシは水生であり、約一週間で組織や器官を幼生型から成体型へと大規模に再構築することで、陸上環境に適応したカエルになる。このメタモルフォーゼの引き金が、たった一つの「甲状腺ホルモン」分子であることは昔から知られている。しかしながら、幼生型から成体型への組織再構築が行われるメカニズムの詳細は、モデル両生類であるアフリカツメガエルやネッタイツメガエルのゲノム配列が解読された今もなお、明らかになっていない。

図1. ネッタイツメガエルのメタモルフォーゼ。約一週間でオタマジャクシからカエルへと華麗に変身。
有尾両生類であるイモリやアホロートルは、大部分の組織や器官、例えば脳や心臓が損傷を受けたとしても、元どおりに再構築することができる。この器官再生は、古くから生物学者を魅了し、世界中の多くの研究者が注目する研究テーマの一つである。細胞の脱分化と組織の再パターン化が大きな鍵を握っているが、そのメカニズムの詳細についても未だ明らかにされていない。
両生類が持つ組織再構築能力のメカニズムの解明は、基礎生物学のみならず再生医学への大きな貢献が期待される。現在、我々のグループでは上述の”Reading”によってもたらされる情報を基に、”Editing”技術を駆使し、両生類の”Reconstructing”能力の謎を解き明かすべく日々努力している。
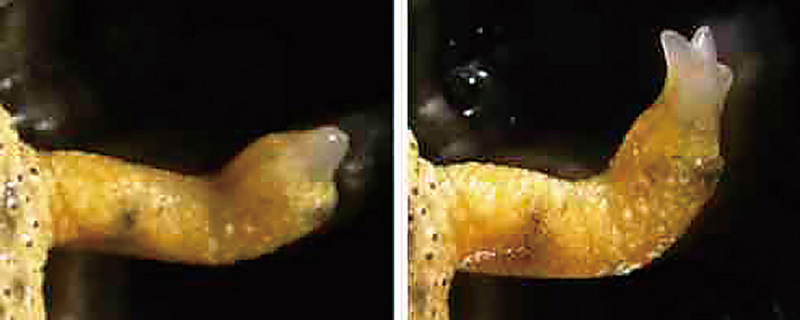
図 2. イベリアトゲイモリの四肢再生。左図のように四肢を失っても、1〜2ヶ月程度で元に戻る。
参考文献
Suzuk,i M., Okumura, A., Chihara, A., Shibata, Y., Endo, T., Teramoto, M., Agata, K., Bronner, M.E., Suzuki, K.T. (2024). Fgf10 mutant newts regenerate normal hindlimbs despite severe developmental defects. Proc. Natl. Acad. Sci. USA 121, e2314911121.
Shibata, Y., Suzuki, M., Hirose, N., Takayama, A., Sanbo, C., Inoue, T., Umesono, Y., Agata, K., Ueno, N., Suzuki, K.T., Mochii, M. (2022). CRISPR/Cas9-based simple transgenesis in Xenopus laevis.Dev. Biol. 489, 76-83.
Suzuki, M., Hayashi, T., Inoue, T., Agata, K., Hirayama, M., Suzuki, M., Shigenobu, S., Takeuchi, T., Yamamoto, T., and Suzuki, K.T. (2018). Cas9 ribonucleoprotein complex allows direct and rapid analysis of coding and noncoding regions of target genes in Pleurodeles waltl development and regeneration. Dev. Biol. 443, 127-136.
Nakade, S., Tsubota, T., Sakane, Y., Kume, S., Sakamoto, N., Obara, M., Daimon, T., Sezutsu, H., Yamamoto, T., Sakuma, T., and Suzuki, K. T. (2014). Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nat. Commun. 5, 5560.








