
基礎生物学研究所





細胞は周囲の環境変化に適応して生存するために、栄養源やストレスといった細胞外からの入力情報を感知し、それらに応答して細胞機能を発現することで細胞の恒常性を維持しています。分子生物学や生化学、細胞生物学の発展に伴い、細胞を構成する分子やそれらの相互作用の理解が進みました。しかし、これらの因子や相互作用がどのようにしてシステムとして協調し、細胞機能を創発しているのかという点については未解明の点が多く残されています。この問題に答えるためには、これまで得られた知見をコンピューター上で再構成し、細胞機能が再現するかどうかを調べることが理想的です。そこで当研究室は、単細胞真核生物の分裂酵母を使い、情報伝達系、代謝、複製-転写-翻訳、細胞形態などを含む「細胞まるごとモデル」を構築することを目指します。モデルに必要な実験データ(タンパク質濃度、解離定数、拡散速度など)を計測し、シミュレーションに使用することで、定量的で予測可能な細胞まるごとモデリングを実装し、細胞機能の創発原理の演繹的な理解を目指します。
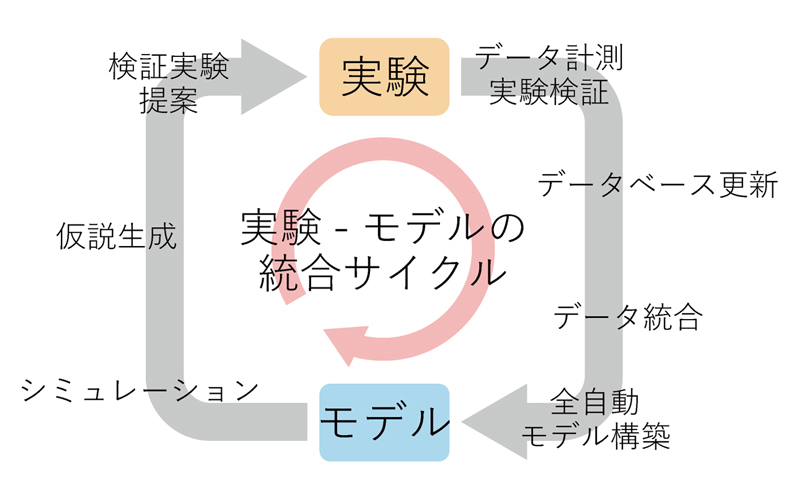
概略図
「細胞まるごとモデル」を構築するためには、情報伝達-細胞周期、代謝、複製-転写-翻訳、細胞形態-細胞状態のモデリングと連関が必要である。マイコプラズマや大腸菌を対象とした原核生物における細胞まるごとモデルは報告されているが、真核生物の細胞まるごとモデルは依然として報告されていない。それは、細胞周期の非線形性、オルガネラ構造、エピジェネティクス、多細胞性などに起因する複雑さと実験データの不足による。そこで、我々は単細胞真核生物のモデル生物である分裂酵母(Schizosaccharomyces pombe)を対象に、実験データの収集とモデル構築-連関を効率良くサイクルすることにより、真核生物における細胞まるごとモデルに挑戦する(概略図)。我々は主に実験データの取得と細胞まるごとモデルを用いた仮説検証を行い、細胞まるごとモデルの構築に関しては共同研究により実施する。
真核生物の細胞まるごとモデルの構築を妨げている要因の一つは、細胞周期の複雑性である。原核生物と異なり、真核生物では細胞周期が秩序だって進行し、それは多くのフィードバック反応を含む非線形なシステムによって成立していることが示唆されている。そこで、当面は分裂酵母の細胞周期を定量的に表現できるモデルを構築することを目指す。当研究室では細胞周期のキーレギュレーターであるCDKやPKAの活性をバイオセンサーにより可視化するツールを開発している(文献2, 4)(図1)。さらに、細胞周期関連因子の細胞内濃度や解離定数、粘性を蛍光相関分光法(FCS)や蛍光相互相関分光法(FCCS)などにより定量化する技術開発も行っている(文献1, 3, 5)。これらの技術を用いて、細胞周期のモデル化に必要な実験データやパラメーター、さらに細胞形態(細胞長、体積、伸長速度、分裂時間、粘弾性等)をイメージングにより取得し、これらを含む数理モデル化を試みる。
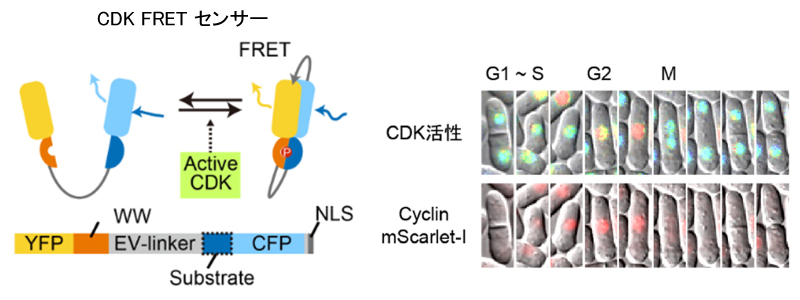
図1. 分裂酵母における細胞周期関連因子の可視化
Cyclin-dependent kinase (CDK) 活性を可視化するFRETバイオセンサー(左)。CDK活性(上段)と内在性のCyclin(下段)の可視化。上段は寒色が低活性、暖色が高活性を示している。