研究の概要
ゲノム比較から多様性理解へ
多様な生物種についてのゲノム解読が進み、それらの比較解析から生物の多様性とそれを生み出す進化プロセスを理解することが可能になりつつあります。当研究室では、特に多様なゲノムが蓄積している微生物のゲノムに着目して、比較ゲノム情報学の立場から、なるべく普遍的な視点でこの問題に取り組もうとしています。すなわち、多数のゲノムを比較して、その間にみられるパターンの共通性と多様性を解析することによって、遺伝子の集合体としてのゲノムの成り立ちを理解し、それによってゲノムの進化過程を推定したり、機能未知遺伝子の機能を推定したりすることを目指します。この目的のため、独自の微生物比較ゲノムデータベースを構築し、これに基づいて比較ゲノム解析の新しいアプローチの開拓を目指した研究を行っています。
微生物ゲノム比較解析システム
直接目に見えない微生物を研究する上で、ゲノム情報はことさらに大きな価値を持つため、すでに多様な微生物のゲノム配列が決定され、その数はなお急速な拡大を続けている。こうした大量のデータに基づく比較ゲノム研究を推進するため、微生物ゲノム比較解析システムMBGDの構築を行っている。特に、比較解析を行う際に必要となるゲノム間の遺伝子対応付け(オーソログ分類)について、効率的なアルゴリズムの開発を行っている。また、オーソログ分類の結果として得られる系統パターン(ある遺伝子が各ゲノム中に存在するかしないかというパターン)や融合タンパク質の存在などから遺伝子の機能推定を行える可能性が指摘されており、大量のデータを活用することにより、その可能性を高めることも目指している。このような解析を効率よく行うため、オーソログ解析に基づいて比較ゲノム解析を行う汎用ワークベンチRECOGの開発を行っている。
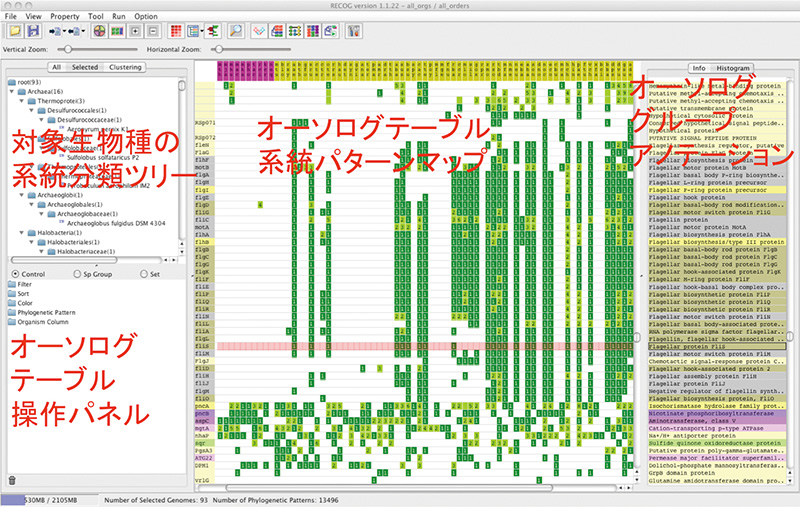
図1. 比較ゲノム解析システムRECOGで表示したオーソログ対応テーブル
近縁ゲノムの比較解析
原核生物の進化においては、祖先から子孫へという垂直的な遺伝情報の流れに加えて、種を超えた水平的な遺伝情報の移動が本質的に重要な役割を果たしていることが知られており、病原性の理解などの応用面からも注目されている。しかし、こうした複雑な微生物のゲノム進化過程を包括して理解するための戦略はまだ確立していない。ゲノム進化過程の詳細な解析は、類縁度の高いゲノムを比較することによって可能になるので、MBGDのデータを活用しつつ、近縁ゲノム比較解析の戦略確立に向けた研究を行っている。特に、ゲノムの垂直的な進化プロセスをまず明確にすることを目指して、近縁ゲノム間で遺伝子の並び順が保存された「コア構造」に着目した研究を進めている。
参考文献
Uchiyama, I., Mihara, M., Nishide, H., Chiba, H., Takayanagi, M., Kawai, M., Takami, H. (2024). MBGD: Microbial Genome Database for Comparative Analysis Featuring Enhanced Functionality to Characterize Gene and Genome Functions Through Large-scale Orthology Analysis. J. Mol. Biol. 437, 168957.
Uchiyama, I., Mihara, M., Nishide, H., Chiba, H., and Kato, M. (2019). MBGD update 2018: microbial genome database based on hierarchical orthology relations covering closely related and distantly related comparisons. Nucleic Acids Res. 47, D382-D389.
Uchiyama, I., Albritton, J., Fukuyo, M., Kojima, K., Yahara, K., and Kobayashi, I. (2016). A novel approach to Helicobacter pylori pan-genome analysis for identification of genomic islands. PLoS One 11, e0159419.
Chiba, H., and Uchiyama, I. (2014). Improvement of domain-level ortholog clustering by optimizing domain-specific sum-of-pairs score. BMC Bioinform. 15, 148.
Uchiyama, I. (2008). Multiple genome alignment for identifying the core structure among moderately related microbial genomes. BMC Genom. 9, 515.
Uchiyama, I. (2006). Hierarchical clustering algorithm for comprehensive orthologous-domain classification in multiple genomes. Nucleic Acids Res. 34, 647-658.






