|
 |
 |
| |
|
 |
|
|
 |
 |
 |
 |
 |
 |
 |
分裂期染色体の構造解析(クロマチンイメージング)に基づく核ゲノム機能の解明
平成17−18年度
X線をもちいた核ゲノム構造の解析
平成17−18年度 |
 |
| 独立行政法人理化学研究所 前島一博 |
| |
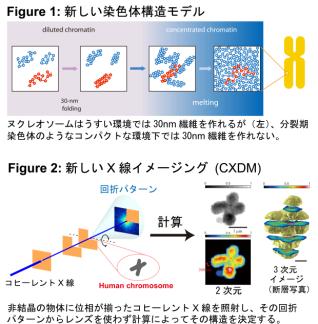
染色体は細胞が分裂する際、複製された遺伝情報(DNA)を2つの娘細胞に正確に分配するために必須な構造体である (6)。それでは、分裂期染色体はどのようにして1本の長いクロマチン繊維から折り畳まれているのだろうか?この問いに答えるために、私たちは、ヒト分裂期細胞を高圧急速凍結し、その細胞の凍結切片を作製して、クライオ電子顕微鏡(CEMOVIS)による観察をおこなった。さらに計算機による画像解析の結果、分裂期染色体中には、30nmクロマチン繊維は検出されず、染色体は11nmのヌクレオソームが不規則に折り畳まれてできていると推定された(2, 6)。
しかしながら、顕微鏡観察は試料中における観察範囲が限定され、内在する規則性構造の全体像を捉えることが非常に困難である。このため、私たちは大型放射光施設SPring-8 でX線小角散乱解析(SAXS)をおこなった。SAXSは、計測したい非結晶試料にX線を照射し、その散乱パターンからその試料に内在する構造や規則性を知る手段である。この解析においても、ヌクレオソームの直径に相当する11nmより大きな構造は、やはり検出されていない(投稿準備中)。このことから私たちは、「染色体内には古くからのモデルが提唱するような明確な階層構造は存在しない」と提唱している (Fig. 1 参照)(2, 6)。
さらに、X線をもちいた新しいナノイメージングとして、播磨理研の西野らと共同で、コヒーレントX線をもちいた新しいX線顕微鏡CXDMを開発した (Fig. 2 参照)。この方法は非結晶の単一の物体に位相が揃ったコヒーレントX線を照射し、その回折(散乱)パターンから、計算によってその構造を決定しようというものである。私たちは、単一のヒト染色体をイメージングし、ヒト染色体の3次元電子密度マップの作製に成功した(1)。今後、生体超分子構造解析への応用が期待されている。
ゲノム機能の理解のためには、細胞核という「入れ物」自体の構造を理解することも必要不可欠である。本特定領域期間中に、私たちは細胞核の構築についても解析を開始し、核膜上の核膜孔が存在しない領域“Pore-free island” を見出した (8, 9)。また、核内ゲノムのorganizationの解析においても、転写の混乱を防ぐためのゲノムの区切りである「インスレーター」に、ゲノムクロマチンを束ねる「コヒーシン複合体」が関与することを明らかにした(4)。このことはゲノムクロマチンの折り畳みが、遺伝子発現制御に直接的に関与することを意味している。
|
| |
発表論文リスト:
- Nishino, Y., Takahashi, Y., Imamoto, N., Ishikawa, T., and Maeshima, K. Three-Dimensional Visualization of a Human Chromosome Using Coherent X-ray Diffraction. Physical Review Letters (2009), 102, 18101 (4 pages) (Cover illustration)
- Eltsov, M., MacLellan, K.M., Maeshima, K., Frangakis, A.S., and Dubochet, J. (Three authors have equal contribution) Analysis of cryo-electron microscopy images does not support the existence of 30-nm chromatin fibers in mitotic chromosomes in situ. Proc. Natl. Acad. Sci. USA (2008) 105, 19732-7.
- Iwai, Y., Ikeda, T., Kojima, T.-M., Yamazaki, Y., Maeshima, K., Imamoto, N., Kobayashi, T., Nebiki, T., Narusawa, T., Pokhil, G.-P. Ion irradiation in liquid of μm3 region for cell surgery. Applied Physics Letters (2008), 92, 23509 (3 pages).
- Wendt, K.S., Yoshida, K., Itoh, T., Bando, M., Koch, B., Schirghuber, E., Tsutsumi, S., Nagae, G., Ishihara, Ko., Mishiro, T., Yahata, K., Imamoto, F., Aburatani, H., Nakao, M., Imamoto, N., Maeshima, K., Shirahige, K., and Peters, J.-M. Cohesin is required for the transcriptional insulator function of CTCF binding sites. Nature (Article) (2008), 451, 796-801.
- Tahara, K., Takagi, M., Ohsugi, M., Sone, T. Nishiumi, F., Maeshima, K., Horiuchi, Y., Tokai-Nishizumi, N., Imamoto, F., Yamamoto, T., Kose, S., and Imamoto, N. Importin-beta and small GTPase Ran mediate chromosome loading of human chromokinesin Kid, Journal of Cell Biology (2008), 180, 493-596.
- Maeshima, K., and Eltsov, M. Packaging the genome: the structure of mitotic chromosomes, Journal of Biochemistry (2008) 143, 145-153. (Cover illustration)
- Yahata, K., Maeshima, K., Sone, T., Ando, T., Okabe, M., Imamoto, N., and Imamoto, F. cHS4 insulator-mediated alleviation of promoter interference during cell based expression of tandemly associated transgenes, Journal of Molecular Biology (2007), 374, 580-90.
- Funakoshi, T., Maeshima, K., Yahata, K., Sugano, S., Imamoto, F., and Imamoto, N. TWO DISTINCT HUMAN POM121 GENES: Requirement for the Formation of Nuclear Pore Complexes, FEBS Letters (2007), 581, 4910-6.
- Maeshima, K., Yahata, K., Sasaki, Y., Nakatomi, R., Tachibana, T., Hashikawa, T., Imamoto, F. and Imamoto, N. Cell cycle dependent dynamics of nuclear pores: pore-free island and lamins, Journal of Cell Science (2006), 119, 4442-4451.
- Maeshima, K., Eltsov, M. and Laemmli, U.K. Chromosome Structure: Improved Immuno-labeling for Electron Microscopy, Chromosoma (2005), 114, 365-75. (Cover illustration)
|
|
 |
 |
 |
 |
 |
 |
 |
| |
|
|
|
|
| |
|
Copyright (c) 2004-2009, nuclear-dynamics. All Rights Reserved. |
|
|
|
|